(3 + (5 * (2^2))) # difícil de leer[1] 233 + 5 * 2^2 # claro, si recuerdas las reglas[1] 233 + 5 * (2^2) # si olvidas algunas reglas, esto podría ayudar[1] 23Toda esta clase se basa en este tutorial de Carpentries.
# Comentario
1 + 100
1 + # y luego [Enter]Las operaciones artimeticas siguen el orden regular de precedencia:
(, )^ o **/*+-(3 + (5 * (2^2))) # difícil de leer[1] 233 + 5 * 2^2 # claro, si recuerdas las reglas[1] 233 + 5 * (2^2) # si olvidas algunas reglas, esto podría ayudar[1] 23Formatos de números:
2 / 10000[1] 2e-045e3 # nota la falta del signo menos aquí[1] 5000sin(1) # función trigonométrica[1] 0.841471log(2) # logaritmo natural[1] 0.6931472log10(2) # logaritmo base 10[1] 0.30103exp(0.5) # e^(1/2)[1] 1.648721Comparaciones
1 == 1[1] TRUE1 != 1[1] FALSE1 != 2[1] TRUE1 < 2[1] TRUE1 <= 2[1] TRUE1 > 2[1] FALSE1 >= 2[1] FALSELos nombres de las variables pueden contener letras, números, guiones bajos y puntos. No pueden comenzar con un número ni contener espacios en absoluto. Diferentes personas usan diferentes convenciones para nombres largos de variables, estos incluyen
x <- 1 / 40
log(x)[1] -3.688879x <- log(x)
x[1] -3.6888791:5[1] 1 2 3 4 52^(1:5)[1] 2 4 8 16 32x <- 1:5
2^x[1] 2 4 8 16 32installed.packages()install.package(nombre_de_paquete)update.packages()remove.packages(nombre_de_paquete)library(nombre_de_paquete)Una mala practica para los proyectos en R es tener un directorio de trabajo con muchos archivos y carpetas.
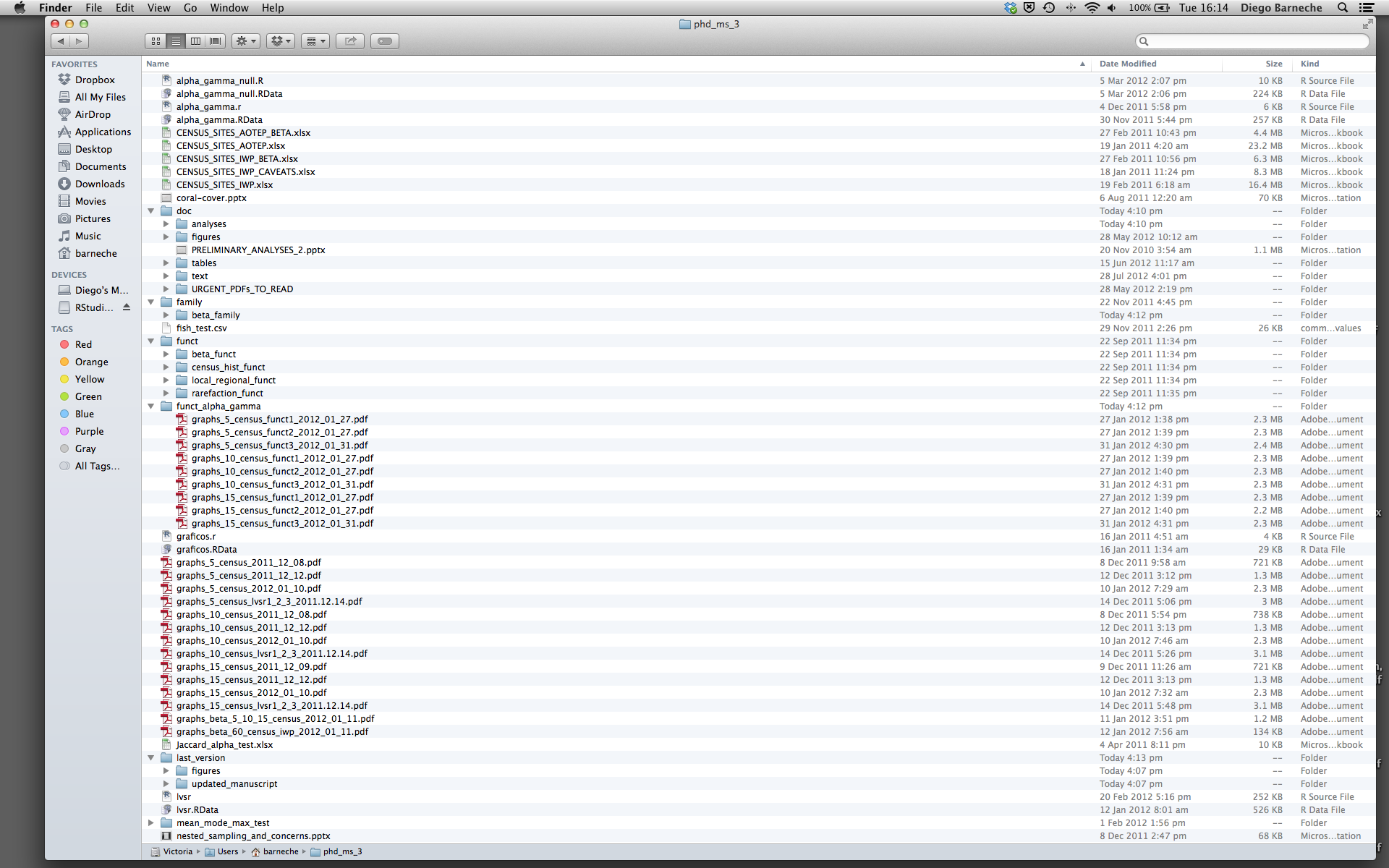
Hay muchas razones de por qué debemos siempre evitar esto:
Un buen diseño del proyecto finalmente hará tu vida más fácil:
Aunque no existe una “mejor” forma de diseñar un proyecto, existen algunos principios generales que deben cumplirse para facilitar su gestión:
install.packages("ProjectTemplate")
library("ProjectTemplate")
create.project("../my_project", merge.strategy = "allow.non.conflict")?función
help(función)
?"<-"vignette()
vignette("nombre_del_paquete")
vigette("nombre_del_paquete", "nombre_del_vignette")??palabra_claveSi no sabes qué función o paquete necesitas usar, utiliza CRAN Task Views es una lista especialmente mantenida de paquetes agrupados en campos. Este puede ser un buen punto de partida.
Si tienes problemas para usar una función, Posiblemente alguien más lo resolvió en Stack Overflow. Puedes buscar usando la etiqueta [r].
install.packages("tidyverse") # Esto se hace solo una vezlibrary(tidyverse)── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
✔ dplyr 1.1.4 ✔ readr 2.1.5
✔ forcats 1.0.0 ✔ stringr 1.5.1
✔ ggplot2 3.5.1 ✔ tibble 3.2.1
✔ lubridate 1.9.3 ✔ tidyr 1.3.1
✔ purrr 1.0.2
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errorsgatos <- data.frame(
color = c("mixto", "negro", "atigrado"),
peso = c(2.1, 5.0, 3.2),
le_gusta_cuerda = c(1, 0, 1)
)
gatos color peso le_gusta_cuerda
1 mixto 2.1 1
2 negro 5.0 0
3 atigrado 3.2 1write_csv(x = gatos, file = "data/gatos-data.csv")gatos2 <- read_csv(file = "data/gatos-data.csv")Rows: 3 Columns: 3
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): color
dbl (2): peso, le_gusta_cuerda
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.gatos$peso[1] 2.1 5.0 3.2gatos$color[1] "mixto" "negro" "atigrado"paste("El color del gato es", gatos$color)[1] "El color del gato es mixto" "El color del gato es negro"
[3] "El color del gato es atigrado"gatos$peso + gatos$colorError in gatos$peso + gatos$color: non-numeric argument to binary operatorclass(gatos$color)[1] "character"class(gatos$peso)[1] "numeric"class(gatos)[1] "data.frame"str(gatos$peso) num [1:3] 2.1 5 3.2vector_coercion <- c("a", TRUE)
str(vector_coercion) chr [1:2] "a" "TRUE"otro_vector_coercion <- c(0, TRUE)
str(otro_vector_coercion) num [1:2] 0 1vector_caracteres <- c("0", "2", "4")
vector_caracteres[1] "0" "2" "4"str(vector_caracteres) chr [1:3] "0" "2" "4"caracteres_coercionados_numerico <- as.numeric(vector_caracteres)
caracteres_coercionados_numerico[1] 0 2 4numerico_coercionado_logico <- as.logical(caracteres_coercionados_numerico)
numerico_coercionado_logico[1] FALSE TRUE TRUEgatos$le_gusta_cuerda[1] 1 0 1class(gatos$le_gusta_cuerda)[1] "numeric"gatos$le_gusta_cuerda <- as.logical(gatos$le_gusta_cuerda)
gatos$le_gusta_cuerda[1] TRUE FALSE TRUEclass(gatos$le_gusta_cuerda)[1] "logical"Otra estructura de datos importante se llama factor.
str(gatos$color) chr [1:3] "mixto" "negro" "atigrado"fct_relevel(gatos$color, c("negro", "mixto", "atigrado"))[1] mixto negro atigrado
Levels: negro mixto atigradoordered(fct_relevel(gatos$color, c("negro", "mixto", "atigrado")))[1] mixto negro atigrado
Levels: negro < mixto < atigradolista <- list(1, "a", TRUE, 1 + 4i)
lista[[1]]
[1] 1
[[2]]
[1] "a"
[[3]]
[1] TRUE
[[4]]
[1] 1+4iotra_lista <- list(title = "Numbers", numbers = 1:10, data = TRUE)
otra_lista$title
[1] "Numbers"
$numbers
[1] 1 2 3 4 5 6 7 8 9 10
$data
[1] TRUEtypeof(gatos)[1] "list"Vemos que los data.frames parecen listas ‘en su cara oculta’ - esto es porque un data.frame es realmente una lista de vectores y factores, como debe ser - para mantener esas columnas que son una combinación de vectores y factores, el data.frame necesita algo más flexible que un vector para poner todas las columnas juntas en una tabla. En otras palabras, un data.frame es una lista especial en la que todos los vectores deben tener la misma longitud.
matrix_example <- matrix(0, ncol = 6, nrow = 3)
matrix_example [,1] [,2] [,3] [,4] [,5] [,6]
[1,] 0 0 0 0 0 0
[2,] 0 0 0 0 0 0
[3,] 0 0 0 0 0 0Prueba estos comandos y diga cuál es la diferencia
matrix(c(4, 1, 9, 5, 10, 7), nrow = 3)matrix(c(4, 9, 10, 1, 5, 7), ncol = 2, byrow = TRUE)matrix(c(4, 9, 10, 1, 5, 7), nrow = 2)matrix(c(4, 1, 9, 5, 10, 7), ncol = 2, byrow = TRUE)Aprendimos que las columnas en un data frame son vectores. Por lo tanto, sabemos que nuestros datos son consistentes con el tipo de dato dentro de esa columna. Si queremos agregar una nueva columna, podemos empezar por crear un nuevo vector:
gatos <- data.frame(
color = c("mixto", "negro", "atigrado"),
peso = c(2.1, 5, 3.2),
legusta_la_cuerda = c(1, 0, 1)
)
gatos$color <- as.factor(gatos$color)
gatosgatos color peso le_gusta_cuerda
1 mixto 2.1 TRUE
2 negro 5.0 FALSE
3 atigrado 3.2 TRUEedad <- c(2, 3, 5)Podemos entonces agregarlo como una columna via:
cbind(gatos, edad) color peso le_gusta_cuerda edad
1 mixto 2.1 TRUE 2
2 negro 5.0 FALSE 3
3 atigrado 3.2 TRUE 5Tenga en cuenta que fallará si tratamos de agregar un vector con un número diferente de entradas que el número de filas en el marco de datos.
edad <- c(2, 3, 5, 12)
cbind(gatos, edad)Error in data.frame(..., check.names = FALSE): arguments imply differing number of rows: 3, 4edad <- c(2, 3)
cbind(gatos, edad)Error in data.frame(..., check.names = FALSE): arguments imply differing number of rows: 3, 2¿Por qué no funcionó? Claro, R quiere ver un elemento en nuestra nueva columna para cada fila de la tabla:
Para que funcione, debemos tener nrow(gatos) = length(edad). Vamos a sobrescribir el contenido de los gatos con nuestro nuevo marco de datos.
edad <- c(2, 3, 5)
gatos <- cbind(gatos, edad)
gatos color peso le_gusta_cuerda edad
1 mixto 2.1 TRUE 2
2 negro 5.0 FALSE 3
3 atigrado 3.2 TRUE 5Ahora, qué tal si agregamos filas, en este caso, la última vez vimos que las filas de un data frame están compuestas por listas:
nueva_fila <- list("carey", 3.3, TRUE, 9)
gatos <- rbind(gatos, nueva_fila)Qué significa el error que nos da R? ‘invalid factor level’ nos dice algo acerca de factores (factors)… pero qué es un factor? Un factor es un tipo de datos en R. Un factor es una categoría (por ejemplo, color) con la que R puede hacer ciertas operaciones. Por ejemplo:
colores <- factor(c("negro", "canela", "canela", "negro"))
levels(colores)[1] "canela" "negro" nlevels(colores)[1] 2Se puede reorganizar el orden de los factores para que en lugar de que aparezcan por orden alfabético sigan el orden elegido por el usuario.
colores ## el orden actual[1] negro canela canela negro
Levels: canela negrocolores <- factor(colores, levels = c("negro", "canela"))
colores # despues de re-organizar[1] negro canela canela negro
Levels: negro canelaLos objetos de la clase factor son otro tipo de datos que debemos usar con cuidado. Cuando R crea un factor, únicamente permite los valores que originalmente estaban allí cuando cargamos los datos. Por ejemplo, en nuestro caso ‘negro’, ‘canela’ y ‘atigrado’. Cualquier categoría nueva que no entre en esas categorías será rechazada (y se conviertirá en NA).
La advertencia (Warning) nos está diciendo que agregamos ‘carey’ a nuestro factor color. Pero los otros valores, 3.3 (de tipo numeric), TRUE (de tipo logical), y 9 (de tipo numeric) se añadieron exitosamente a peso, le_gusta_cuerda, y edad, respectivamente, dado que esos valores no son de tipo factor. Para añadir una nueva categoría ‘carey’ al data frame gatos en la columna color, debemos agregar explícitamente a ‘carey’ como un nuevo nivel (level) en el factor:
levels(gatos$color)NULLlevels(gatos$color) <- c(levels(gatos$color), "carey")
gatos <- rbind(gatos, list("carey", 3.3, TRUE, 9))Warning in `[<-.factor`(`*tmp*`, ri, value = structure(c("mixto", "negro", :
invalid factor level, NA generatedDe manera alternativa, podemos cambiar la columna a tipo character. En este caso, perdemos las categorías, pero a partir de ahora podemos incorporar cualquier palabra a la columna, sin problemas con los niveles del factor.
str(gatos)'data.frame': 5 obs. of 4 variables:
$ color : Factor w/ 1 level "carey": NA NA NA 1 1
$ peso : num 2.1 5 3.2 3.3 3.3
$ le_gusta_cuerda: logi TRUE FALSE TRUE TRUE TRUE
$ edad : num 2 3 5 9 9gatos$color <- as.character(gatos$color)
str(gatos)'data.frame': 5 obs. of 4 variables:
$ color : chr NA NA NA "carey" ...
$ peso : num 2.1 5 3.2 3.3 3.3
$ le_gusta_cuerda: logi TRUE FALSE TRUE TRUE TRUE
$ edad : num 2 3 5 9 9x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c("a", "b", "c", "d", "e")
x a b c d e
5.4 6.2 7.1 4.8 7.5 x[1] a
5.4 x[4] d
4.8 La función parentesis cuadrado [] devuelve el \(n\)-ésimo elemento de un vector.
x[c(1, 3)] a c
5.4 7.1 O podemos tomar un rango del vector:
x[1:4] a b c d
5.4 6.2 7.1 4.8 x[6]<NA>
NA La númeración en R comienza en 1, no en 0.
x[-2] a c d e
5.4 7.1 4.8 7.5 x[c(-1, -5)] # o bien x[-c(1,5)] b c d
6.2 7.1 4.8 ¿Por qué no funciona esto?
x[-1:3]Error in x[-1:3]: only 0's may be mixed with negative subscriptsx[-(1:3)] d e
4.8 7.5 Para quitar los elementos de un vector, será necesario que asignes el resultado de vuelta a la variable:
x <- x[-4]
x a b c e
5.4 6.2 7.1 7.5 Con este conjunto
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c("a", "b", "c", "d", "e")
print(x) a b c d e
5.4 6.2 7.1 4.8 7.5 # podemos nombrar un vector en la misma línea
x <- c(a = 5.4, b = 6.2, c = 7.1, d = 4.8, e = 7.5)
print(x) a b c d e
5.4 6.2 7.1 4.8 7.5 x[c("a", "c")] a c
5.4 7.1 Esta forma es mucho más segura para hacer subconjuntos: las posiciones de muchos elementos pueden cambiar a menudo cuando estamos creando una cadena de subconjuntos, ¡pero los nombres siempre permanecen iguales!
También podemos usar un vector con elementos lógicos para hacer subconjuntos:
x[c(FALSE, FALSE, TRUE, FALSE, TRUE)] c e
7.1 7.5 Escribe un comando para crear el subconjunto de valores de x que sean mayores a 4 pero menores que 7.
x_subset <- x[x < 7 & x > 4]
print(x_subset) a b d
5.4 6.2 4.8 Se pueden hacer subconjuntos de listas:
xlist <- list(a = "Cadena de texto", b = 1:10, data = head(mtcars))
str(xlist)List of 3
$ a : chr "Cadena de texto"
$ b : int [1:10] 1 2 3 4 5 6 7 8 9 10
$ data:'data.frame': 6 obs. of 11 variables:
..$ mpg : num [1:6] 21 21 22.8 21.4 18.7 18.1
..$ cyl : num [1:6] 6 6 4 6 8 6
..$ disp: num [1:6] 160 160 108 258 360 225
..$ hp : num [1:6] 110 110 93 110 175 105
..$ drat: num [1:6] 3.9 3.9 3.85 3.08 3.15 2.76
..$ wt : num [1:6] 2.62 2.88 2.32 3.21 3.44 ...
..$ qsec: num [1:6] 16.5 17 18.6 19.4 17 ...
..$ vs : num [1:6] 0 0 1 1 0 1
..$ am : num [1:6] 1 1 1 0 0 0
..$ gear: num [1:6] 4 4 4 3 3 3
..$ carb: num [1:6] 4 4 1 1 2 1xlist[1]$a
[1] "Cadena de texto"Para extraer elementos individuales de la lista, tendrás que hacer uso de la función doble corchete: [[.
xlist[[1]][1] "Cadena de texto"La función $ es una manera abreviada para extraer elementos por nombre:
xlist$data mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1# if
if (la condición es verdad) {
realizar una acción
}
# if ... else
if (la condición es verdad) {
realizar una acción
} else { # es decir, si la condición es falsa,
realizar una acción alternativa
}x <- 8
if (x >= 10) {
print("x es mayor o igual a 10")
} else if (x > 5) {
print("x es mayor a 5, pero menor a 10")
} else{
print("x es menor a 5")
}[1] "x es mayor a 5, pero menor a 10"Recuerden que las instrucciones if evalúan falso o verdadero.
x <- 4 == 3
if (x) {
"4 igual a 3"
} else {
"4 no es igual a 3"
}[1] "4 no es igual a 3"Como podemos observar se muestra el mensaje es igual porque el vector x es FALSE
x <- 4 == 3
x[1] FALSEfor(iterador in conjunto de valores){
haz alguna acción
}output_vector <- c()
for(i in 1:5){
for(j in c('a', 'b', 'c', 'd', 'e')){
temp_output <- paste(i, j)
output_vector <- c(output_vector, temp_output)
}
}
output_vector [1] "1 a" "1 b" "1 c" "1 d" "1 e" "2 a" "2 b" "2 c" "2 d" "2 e" "3 a" "3 b"
[13] "3 c" "3 d" "3 e" "4 a" "4 b" "4 c" "4 d" "4 e" "5 a" "5 b" "5 c" "5 d"
[25] "5 e"Esta es una versión un poco más eficiente
output_matrix <- matrix(nrow=5, ncol=5)
j_vector <- c('a', 'b', 'c', 'd', 'e')
for(i in 1:5){
for(j in 1:5){
temp_j_value <- j_vector[j]
temp_output <- paste(i, temp_j_value)
output_matrix[i, j] <- temp_output
}
}
output_vector2 <- as.vector(output_matrix)
output_vector2 [1] "1 a" "2 a" "3 a" "4 a" "5 a" "1 b" "2 b" "3 b" "4 b" "5 b" "1 c" "2 c"
[13] "3 c" "4 c" "5 c" "1 d" "2 d" "3 d" "4 d" "5 d" "1 e" "2 e" "3 e" "4 e"
[25] "5 e"# Crear la matriz inicial
output_matrix <- matrix(nrow=5, ncol=5)
# Vector de letras
j_vector <- c('a', 'b', 'c', 'd', 'e')
sapply(
X = j_vector,
FUN = function(j) {
sapply(1:5, function(i) {
paste(i, j)
})
}
) a b c d e
[1,] "1 a" "1 b" "1 c" "1 d" "1 e"
[2,] "2 a" "2 b" "2 c" "2 d" "2 e"
[3,] "3 a" "3 b" "3 c" "3 d" "3 e"
[4,] "4 a" "4 b" "4 c" "4 d" "4 e"
[5,] "5 a" "5 b" "5 c" "5 d" "5 e"# Vector de números y letras
i_vector <- 1:5
j_vector <- c('a', 'b', 'c', 'd', 'e')
# Usar lapply para crear combinaciones de i y j
output_list <- lapply(i_vector, function(i) {
sapply(j_vector, function(j) paste(i, j))
})
# Convertir la lista en un vector
output_vector_lapply <- unlist(output_list)
output_vector_lapply a b c d e a b c d e a b c
"1 a" "1 b" "1 c" "1 d" "1 e" "2 a" "2 b" "2 c" "2 d" "2 e" "3 a" "3 b" "3 c"
d e a b c d e a b c d e
"3 d" "3 e" "4 a" "4 b" "4 c" "4 d" "4 e" "5 a" "5 b" "5 c" "5 d" "5 e" while(mientras esta condición es verdad){
haz algo
}z <- 1
while(z > 0.1){
z <- runif(1)
print(z)
}