# Instalación y carga de Tidyverse
install.packages("tidyverse")6 Tidyverse
Primero, instalemos y carguemos el paquete Tidyverse.
library(tidyverse)
library(cowplot)
Attaching package: 'cowplot'The following object is masked from 'package:lubridate':
stamp6.1 Carga de los datos
Vamos a cargar el dataset de Gapminder desde una URL.
# Cargar datos de Gapminder
gapminder <- read_csv("https://raw.githubusercontent.com/resbaz/r-novice-gapminder-files/master/data/gapminder-FiveYearData.csv")Rows: 1704 Columns: 6
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (2): country, continent
dbl (4): year, pop, lifeExp, gdpPercap
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.6.2 Visualización de los datos con ggplot2
Comencemos visualizando los datos con ggplot2.

Veamos la relación entre el PIB per cápita y la esperanza de vida.
6.2.1 Gráfico básico de dispersión
# Gráfico básico de dispersión con ggplot2
gapminder %>%
ggplot(aes(x = gdpPercap, y = lifeExp)) +
geom_point() +
labs(
title = "Esperanza de vida vs PIB per cápita",
x = "PIB per cápita",
y = "Esperanza de vida"
) +
theme_minimal()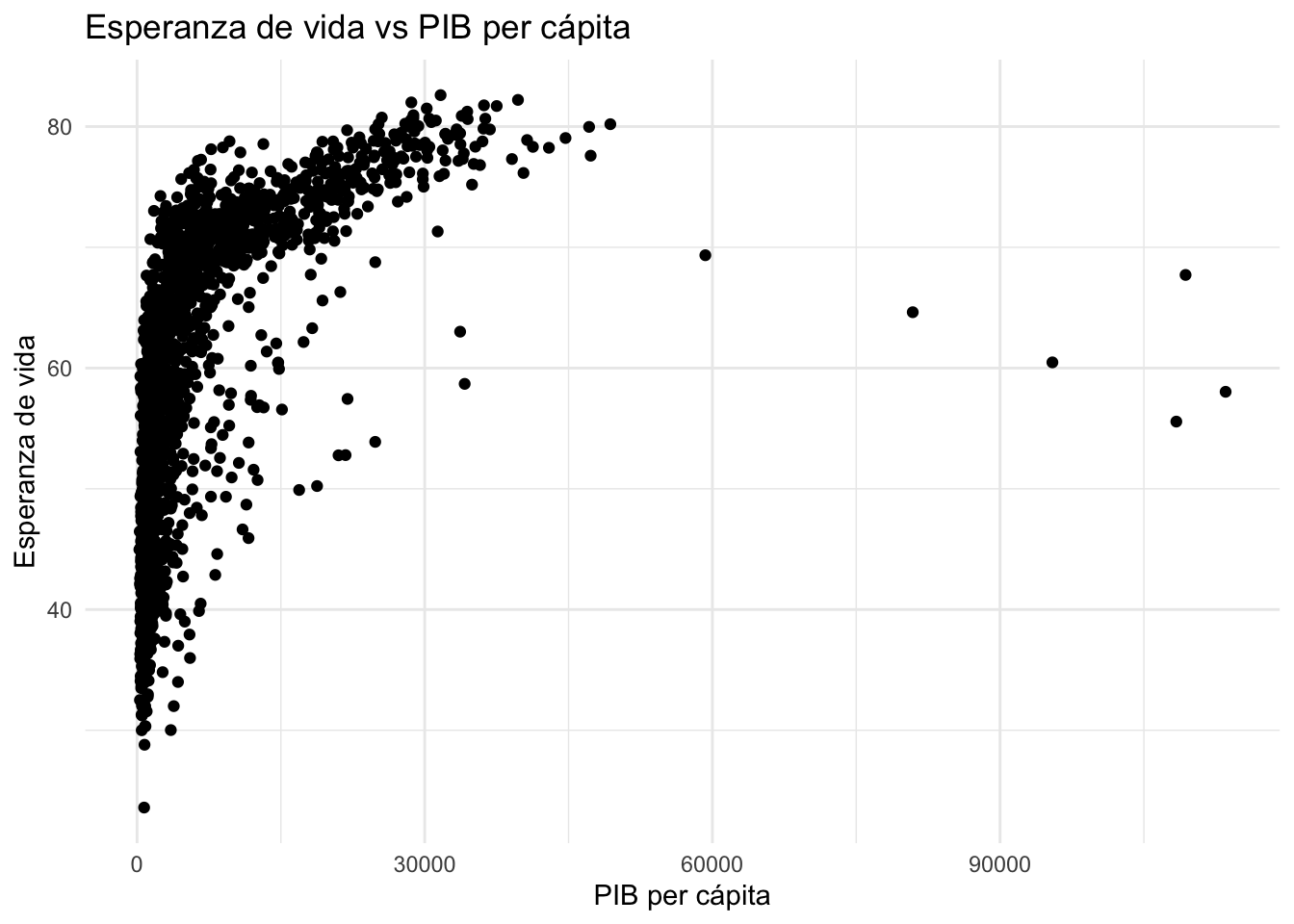
6.2.2 Añadir color por continente
Mejoremos el gráfico añadiendo color para distinguir los continentes.
# Añadir color por continente
gapminder %>%
ggplot(aes(x = gdpPercap, y = lifeExp, color = continent)) +
geom_point() +
labs(
title = "Esperanza de vida vs PIB per cápita por continente",
x = "PIB per cápita",
y = "Esperanza de vida"
) +
scale_x_log10() + # Escala logarítmica para PIB
theme_cowplot()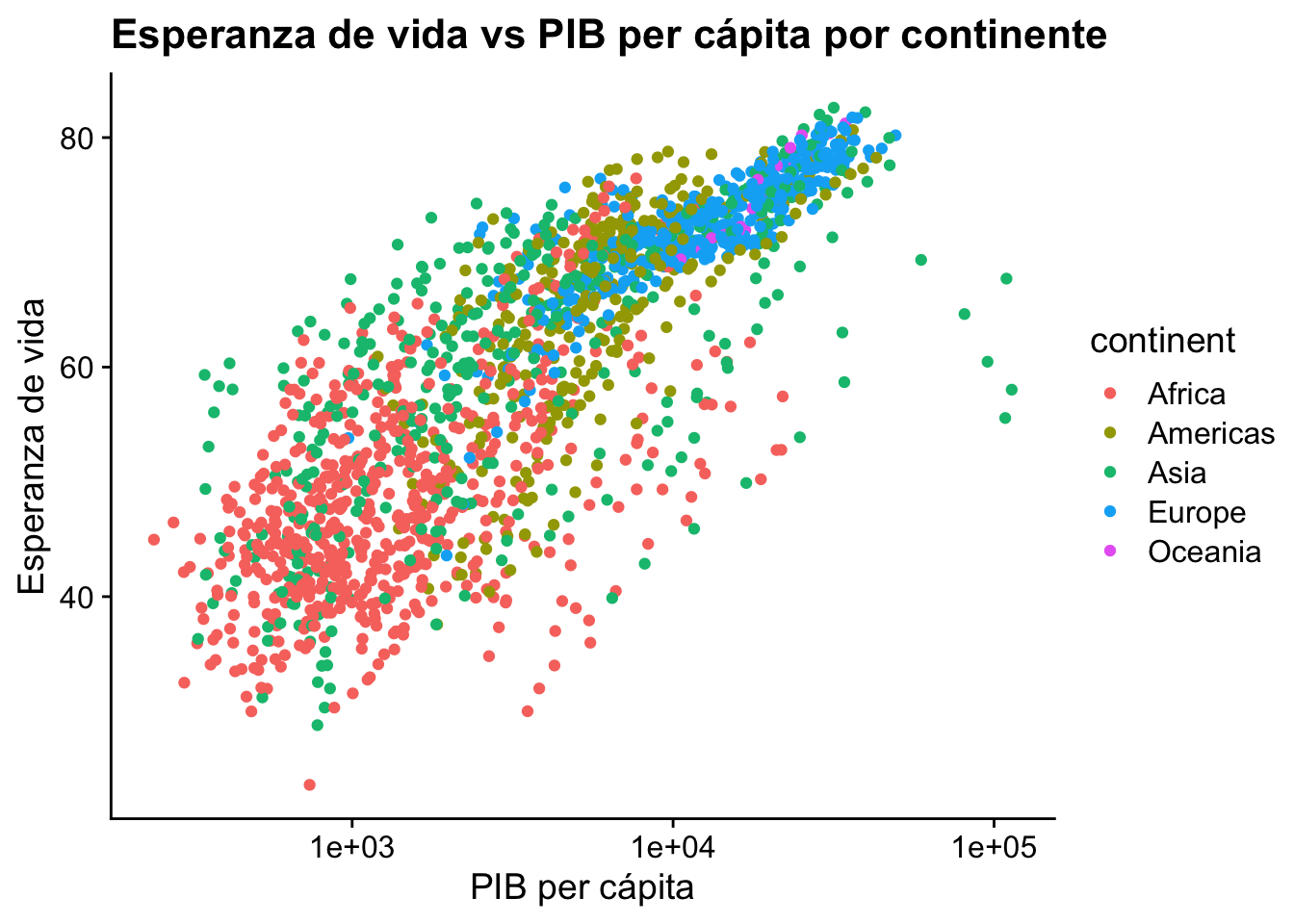
6.2.3 Añadir líneas de tendencia por continente
Para analizar las tendencias dentro de los continentes, añadimos líneas de regresión lineal para cada continente.
# Añadir líneas de tendencia
gapminder %>%
ggplot(aes(x = gdpPercap, y = lifeExp, color = continent)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
labs(
title = "Esperanza de vida vs PIB per cápita por continente con líneas de tendencia",
x = "PIB per cápita",
y = "Esperanza de vida"
) +
scale_x_log10() +
theme_minimal()`geom_smooth()` using formula = 'y ~ x'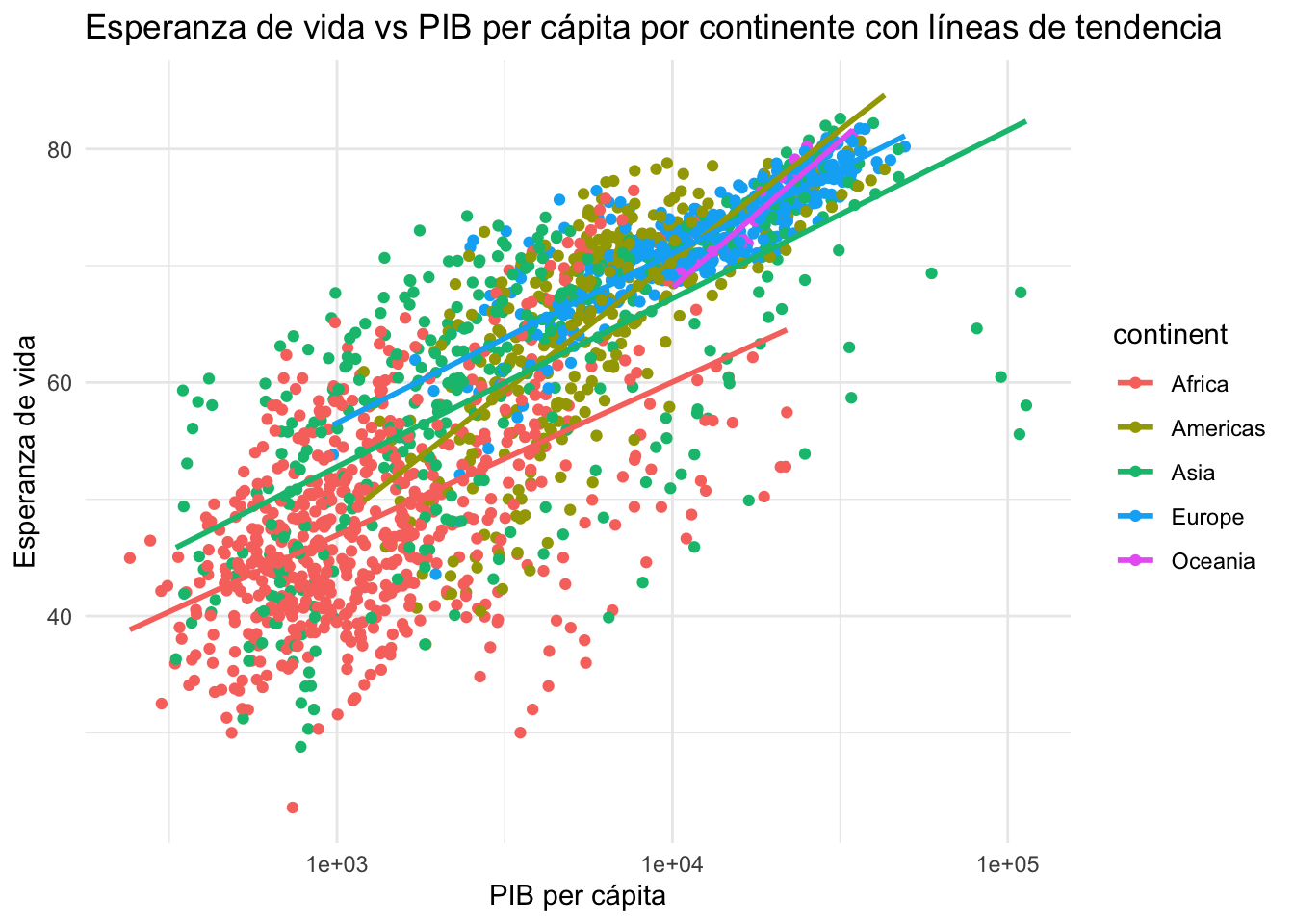
6.2.4 Crear gráficos facetados
Otra poderosa característica de ggplot2 es la posibilidad de crear gráficos “facetados”, es decir, dividir el gráfico en subgráficos basados en una variable, como los continentes.
# Crear gráficos facetados por continente
gapminder %>%
ggplot(aes(x = gdpPercap, y = lifeExp)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
facet_wrap(~continent) +
labs(
title = "Esperanza de vida vs PIB per cápita por continente",
x = "PIB per cápita",
y = "Esperanza de vida"
) +
scale_x_log10(labels = scales::number) +
cowplot::theme_cowplot()`geom_smooth()` using formula = 'y ~ x'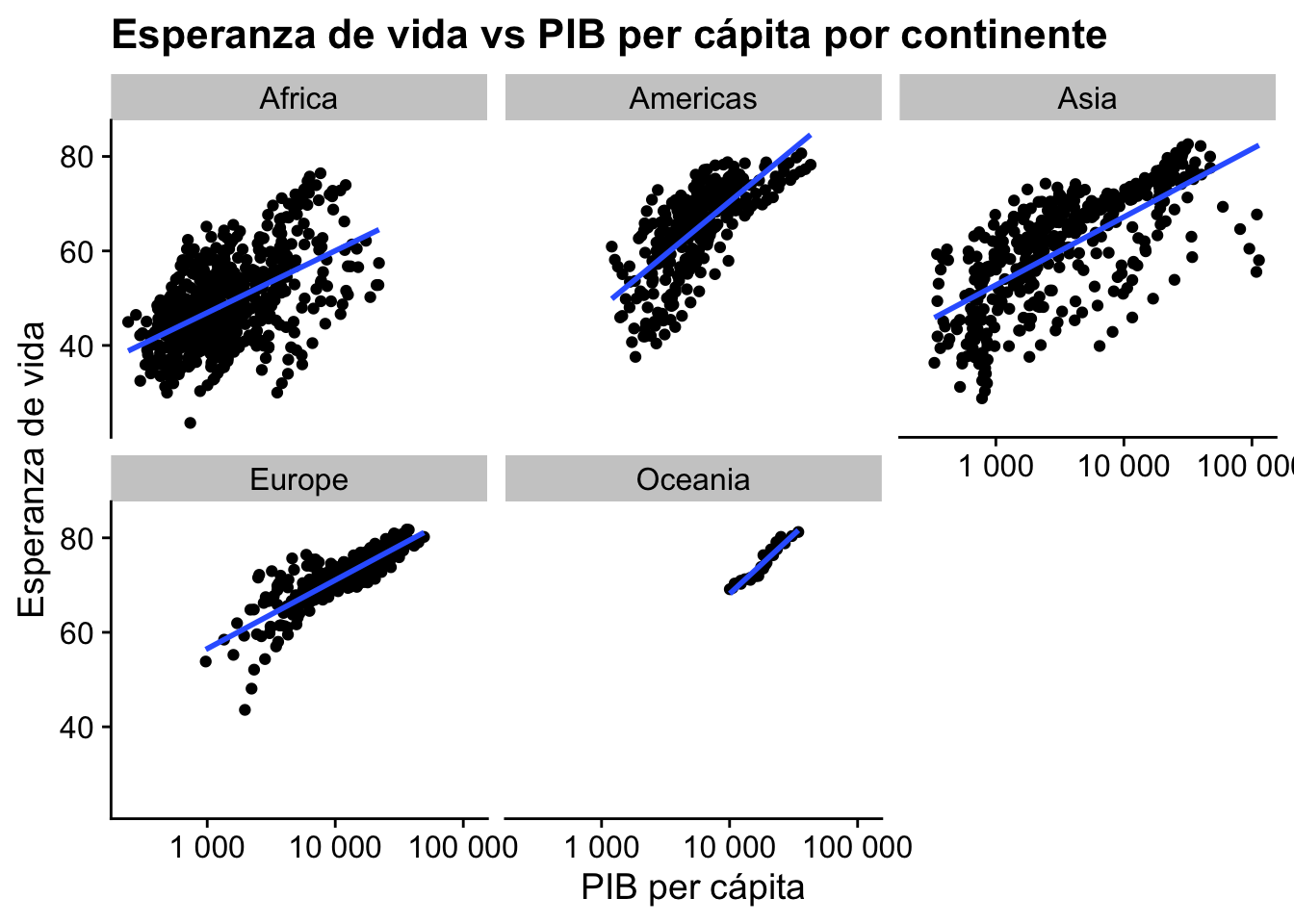
6.3 Tipos de gráficos en ggplot2
Supongamos que tenemos este conjunto de datos llamado: Datos de Lending Club.
- Miles de préstamos realizados a través de Lending Club, una plataforma que permite a individuos prestar a otros individuos.
- Los préstamos varían en facilidad de obtención, dependiendo de la capacidad aparente de pago del prestatario.
- Los datos incluyen solo préstamos otorgados.
library(openintro)Loading required package: airportsLoading required package: cherryblossomLoading required package: usdataglimpse(loans_full_schema)Rows: 10,000
Columns: 55
$ emp_title <chr> "global config engineer ", "warehouse…
$ emp_length <dbl> 3, 10, 3, 1, 10, NA, 10, 10, 10, 3, 1…
$ state <fct> NJ, HI, WI, PA, CA, KY, MI, AZ, NV, I…
$ homeownership <fct> MORTGAGE, RENT, RENT, RENT, RENT, OWN…
$ annual_income <dbl> 90000, 40000, 40000, 30000, 35000, 34…
$ verified_income <fct> Verified, Not Verified, Source Verifi…
$ debt_to_income <dbl> 18.01, 5.04, 21.15, 10.16, 57.96, 6.4…
$ annual_income_joint <dbl> NA, NA, NA, NA, 57000, NA, 155000, NA…
$ verification_income_joint <fct> , , , , Verified, , Not Verified, , ,…
$ debt_to_income_joint <dbl> NA, NA, NA, NA, 37.66, NA, 13.12, NA,…
$ delinq_2y <int> 0, 0, 0, 0, 0, 1, 0, 1, 1, 0, 0, 0, 0…
$ months_since_last_delinq <int> 38, NA, 28, NA, NA, 3, NA, 19, 18, NA…
$ earliest_credit_line <dbl> 2001, 1996, 2006, 2007, 2008, 1990, 2…
$ inquiries_last_12m <int> 6, 1, 4, 0, 7, 6, 1, 1, 3, 0, 4, 4, 8…
$ total_credit_lines <int> 28, 30, 31, 4, 22, 32, 12, 30, 35, 9,…
$ open_credit_lines <int> 10, 14, 10, 4, 16, 12, 10, 15, 21, 6,…
$ total_credit_limit <int> 70795, 28800, 24193, 25400, 69839, 42…
$ total_credit_utilized <int> 38767, 4321, 16000, 4997, 52722, 3898…
$ num_collections_last_12m <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ num_historical_failed_to_pay <int> 0, 1, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0…
$ months_since_90d_late <int> 38, NA, 28, NA, NA, 60, NA, 71, 18, N…
$ current_accounts_delinq <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ total_collection_amount_ever <int> 1250, 0, 432, 0, 0, 0, 0, 0, 0, 0, 0,…
$ current_installment_accounts <int> 2, 0, 1, 1, 1, 0, 2, 2, 6, 1, 2, 1, 2…
$ accounts_opened_24m <int> 5, 11, 13, 1, 6, 2, 1, 4, 10, 5, 6, 7…
$ months_since_last_credit_inquiry <int> 5, 8, 7, 15, 4, 5, 9, 7, 4, 17, 3, 4,…
$ num_satisfactory_accounts <int> 10, 14, 10, 4, 16, 12, 10, 15, 21, 6,…
$ num_accounts_120d_past_due <int> 0, 0, 0, 0, 0, 0, 0, NA, 0, 0, 0, 0, …
$ num_accounts_30d_past_due <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ num_active_debit_accounts <int> 2, 3, 3, 2, 10, 1, 3, 5, 11, 3, 2, 2,…
$ total_debit_limit <int> 11100, 16500, 4300, 19400, 32700, 272…
$ num_total_cc_accounts <int> 14, 24, 14, 3, 20, 27, 8, 16, 19, 7, …
$ num_open_cc_accounts <int> 8, 14, 8, 3, 15, 12, 7, 12, 14, 5, 8,…
$ num_cc_carrying_balance <int> 6, 4, 6, 2, 13, 5, 6, 10, 14, 3, 5, 3…
$ num_mort_accounts <int> 1, 0, 0, 0, 0, 3, 2, 7, 2, 0, 2, 3, 3…
$ account_never_delinq_percent <dbl> 92.9, 100.0, 93.5, 100.0, 100.0, 78.1…
$ tax_liens <int> 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ public_record_bankrupt <int> 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0…
$ loan_purpose <fct> moving, debt_consolidation, other, de…
$ application_type <fct> individual, individual, individual, i…
$ loan_amount <int> 28000, 5000, 2000, 21600, 23000, 5000…
$ term <dbl> 60, 36, 36, 36, 36, 36, 60, 60, 36, 3…
$ interest_rate <dbl> 14.07, 12.61, 17.09, 6.72, 14.07, 6.7…
$ installment <dbl> 652.53, 167.54, 71.40, 664.19, 786.87…
$ grade <fct> C, C, D, A, C, A, C, B, C, A, C, B, C…
$ sub_grade <fct> C3, C1, D1, A3, C3, A3, C2, B5, C2, A…
$ issue_month <fct> Mar-2018, Feb-2018, Feb-2018, Jan-201…
$ loan_status <fct> Current, Current, Current, Current, C…
$ initial_listing_status <fct> whole, whole, fractional, whole, whol…
$ disbursement_method <fct> Cash, Cash, Cash, Cash, Cash, Cash, C…
$ balance <dbl> 27015.86, 4651.37, 1824.63, 18853.26,…
$ paid_total <dbl> 1999.330, 499.120, 281.800, 3312.890,…
$ paid_principal <dbl> 984.14, 348.63, 175.37, 2746.74, 1569…
$ paid_interest <dbl> 1015.19, 150.49, 106.43, 566.15, 754.…
$ paid_late_fees <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…Seleccionamos las siguientes variables
loans <- loans_full_schema %>%
select(
loan_amount, interest_rate, term, grade,
state, annual_income, homeownership, debt_to_income
)
glimpse(loans)Rows: 10,000
Columns: 8
$ loan_amount <int> 28000, 5000, 2000, 21600, 23000, 5000, 24000, 20000, 20…
$ interest_rate <dbl> 14.07, 12.61, 17.09, 6.72, 14.07, 6.72, 13.59, 11.99, 1…
$ term <dbl> 60, 36, 36, 36, 36, 36, 60, 60, 36, 36, 60, 60, 36, 60,…
$ grade <fct> C, C, D, A, C, A, C, B, C, A, C, B, C, B, D, D, D, F, E…
$ state <fct> NJ, HI, WI, PA, CA, KY, MI, AZ, NV, IL, IL, FL, SC, CO,…
$ annual_income <dbl> 90000, 40000, 40000, 30000, 35000, 34000, 35000, 110000…
$ homeownership <fct> MORTGAGE, RENT, RENT, RENT, RENT, OWN, MORTGAGE, MORTGA…
$ debt_to_income <dbl> 18.01, 5.04, 21.15, 10.16, 57.96, 6.46, 23.66, 16.19, 3…Podemos ver sus tipos:
| Variable | Tipo |
|---|---|
loan_amount |
Numérica, continua |
interest_rate |
Numérica, continua |
term |
Numérica, discreta |
grade |
Categórica, ordinal |
state |
Categórica, no ordinal |
annual_income |
Numérica, continua |
homeownership |
Categórica, no ordinal |
debt_to_income |
Numérica, continua |
Nuestro objetivo es identificar diferentes patrones como:
- Forma:
- Sesgada a la derecha, sesgada a la izquierda, simétrica.
- Modality: unimodal, bimodal, multimodal, uniforme.
- Centro: Media, mediana, moda.
- Dispersión: Rango, desviación estándar, rango intercuartílico (IQR).
- Observaciones inusuales.
6.3.1 Histograma
ggplot(loans, aes(x = loan_amount)) +
geom_histogram(color = "white")`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.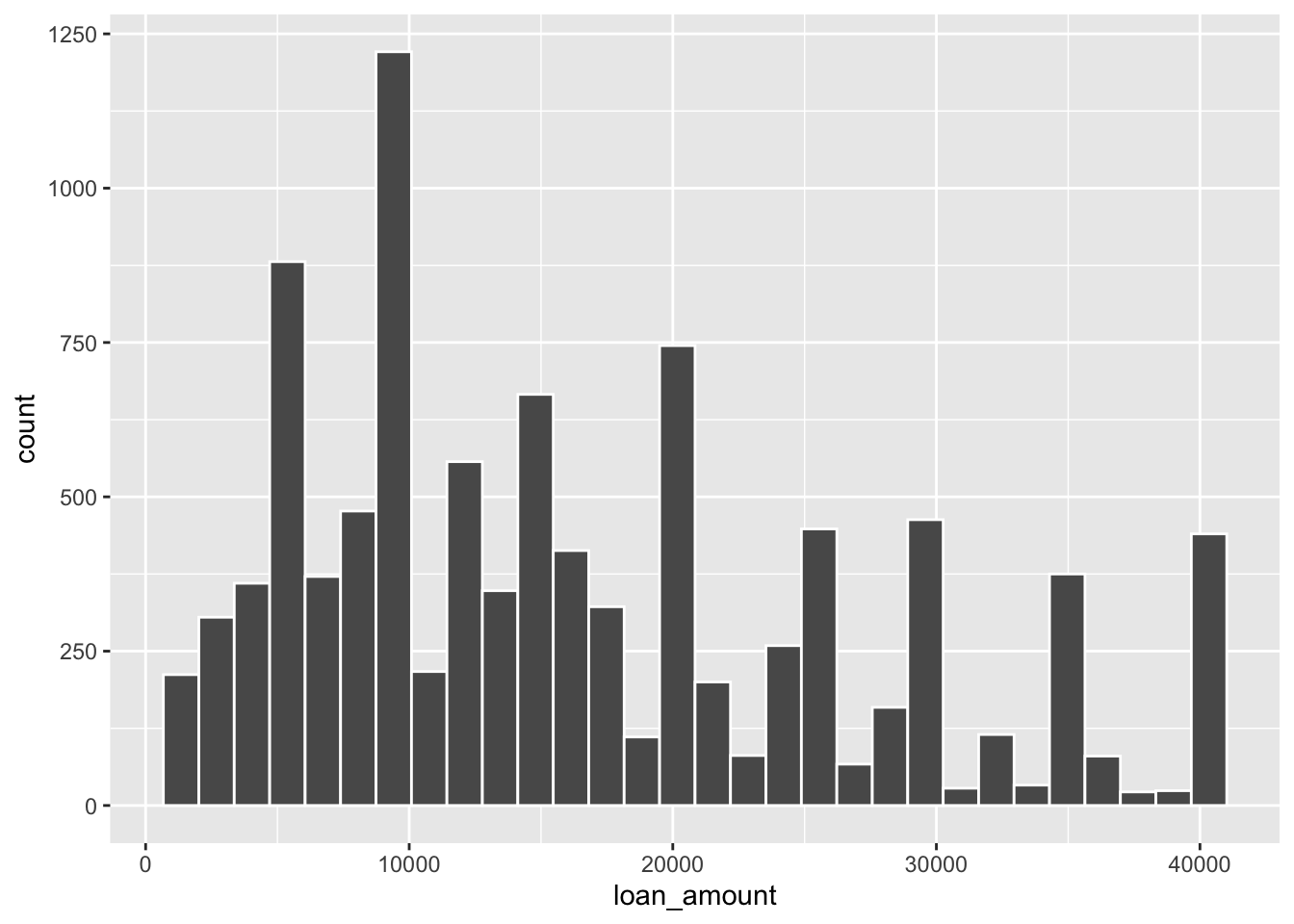
Cambiando el ancho de los bins
ggplot(loans, aes(x = loan_amount)) +
geom_histogram(binwidth = 10)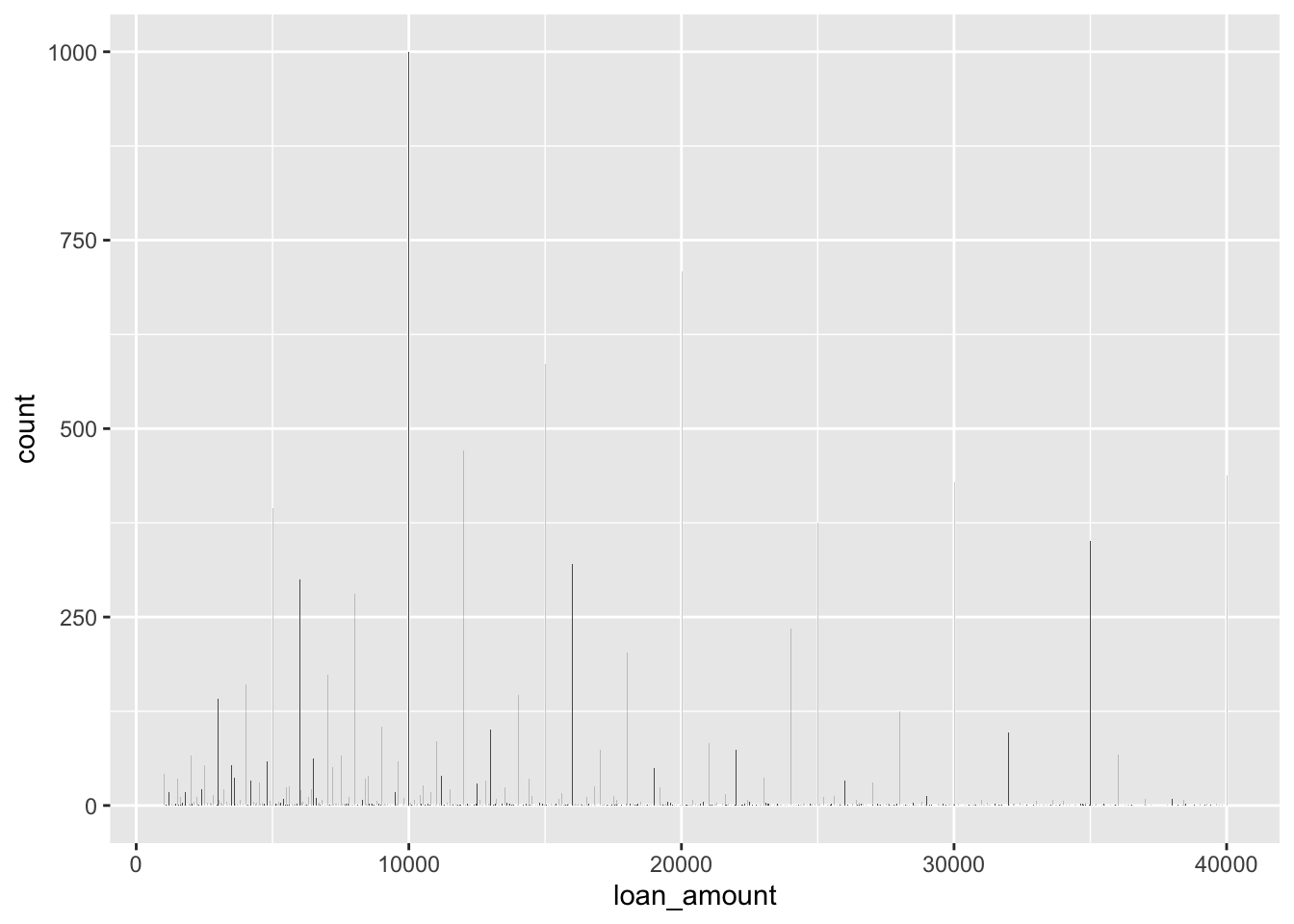
ggplot(loans, aes(x = loan_amount)) +
geom_histogram(binwidth = 5000)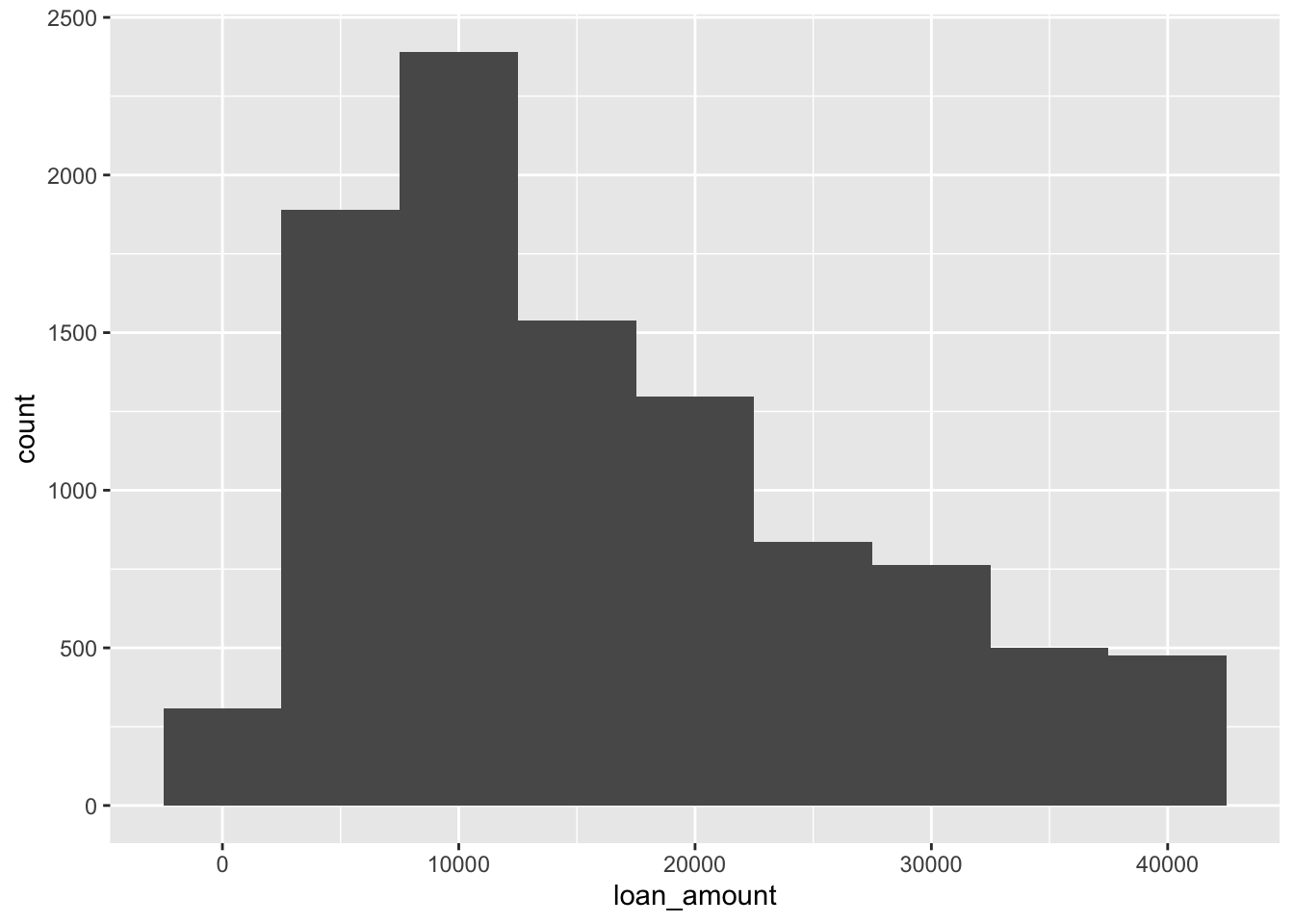
ggplot(loans, aes(x = loan_amount)) +
geom_histogram(binwidth = 20000)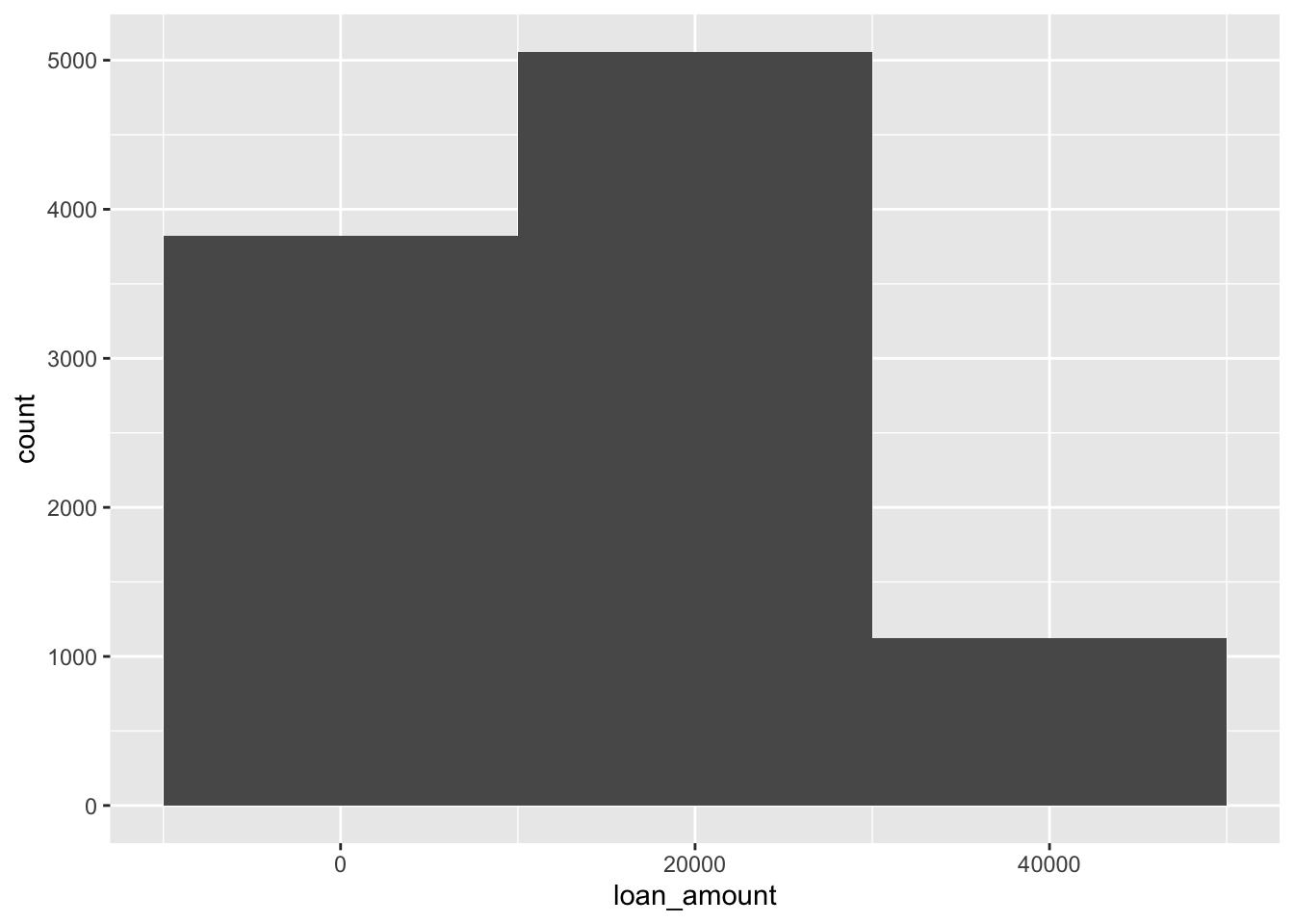
Personalización de histogramas
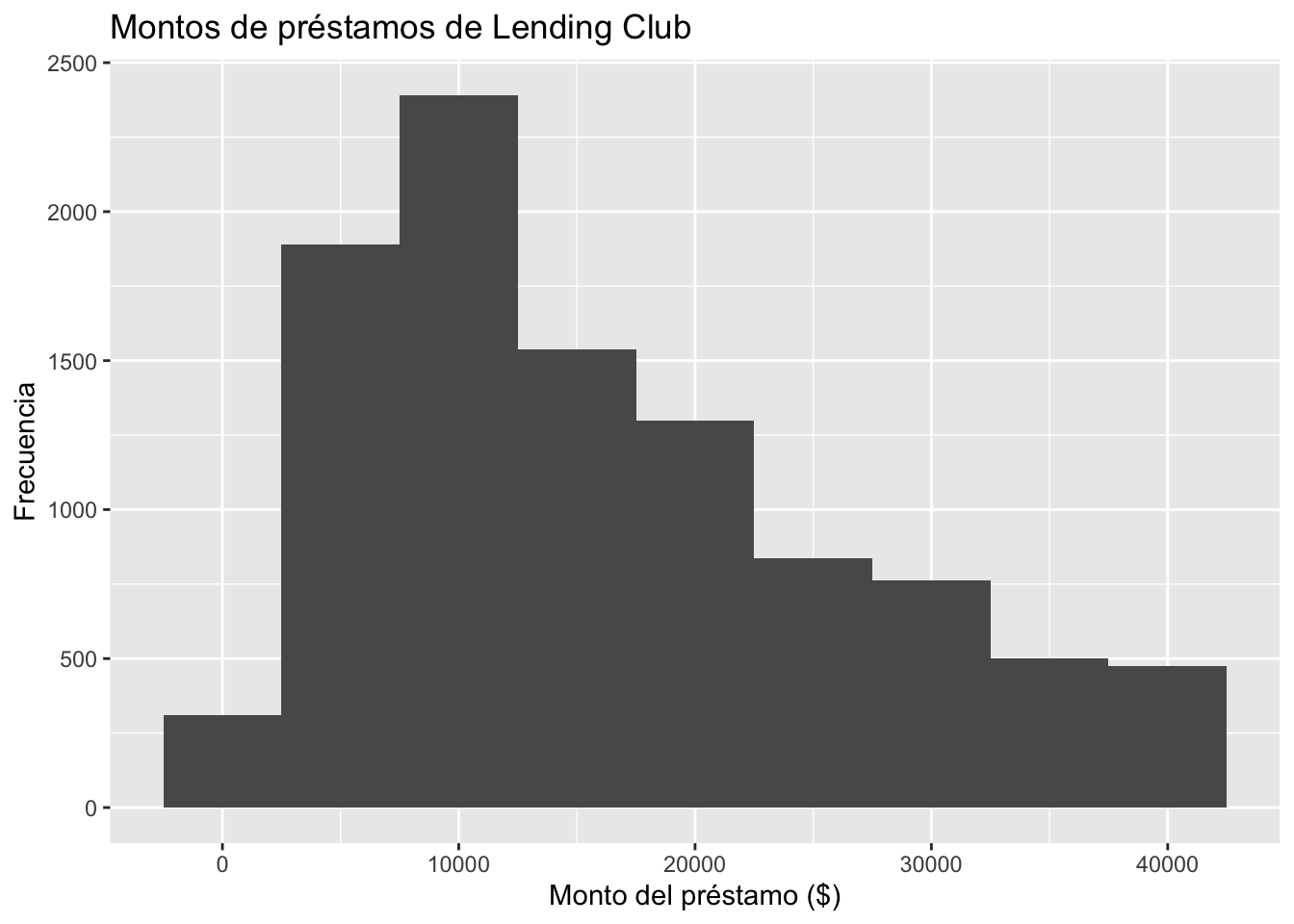
ggplot(loans, aes(x = loan_amount)) +
geom_histogram(binwidth = 5000) +
labs(
x = "Monto del préstamo ($)",
y = "Frecuencia",
title = "Montos de préstamos de Lending Club"
)
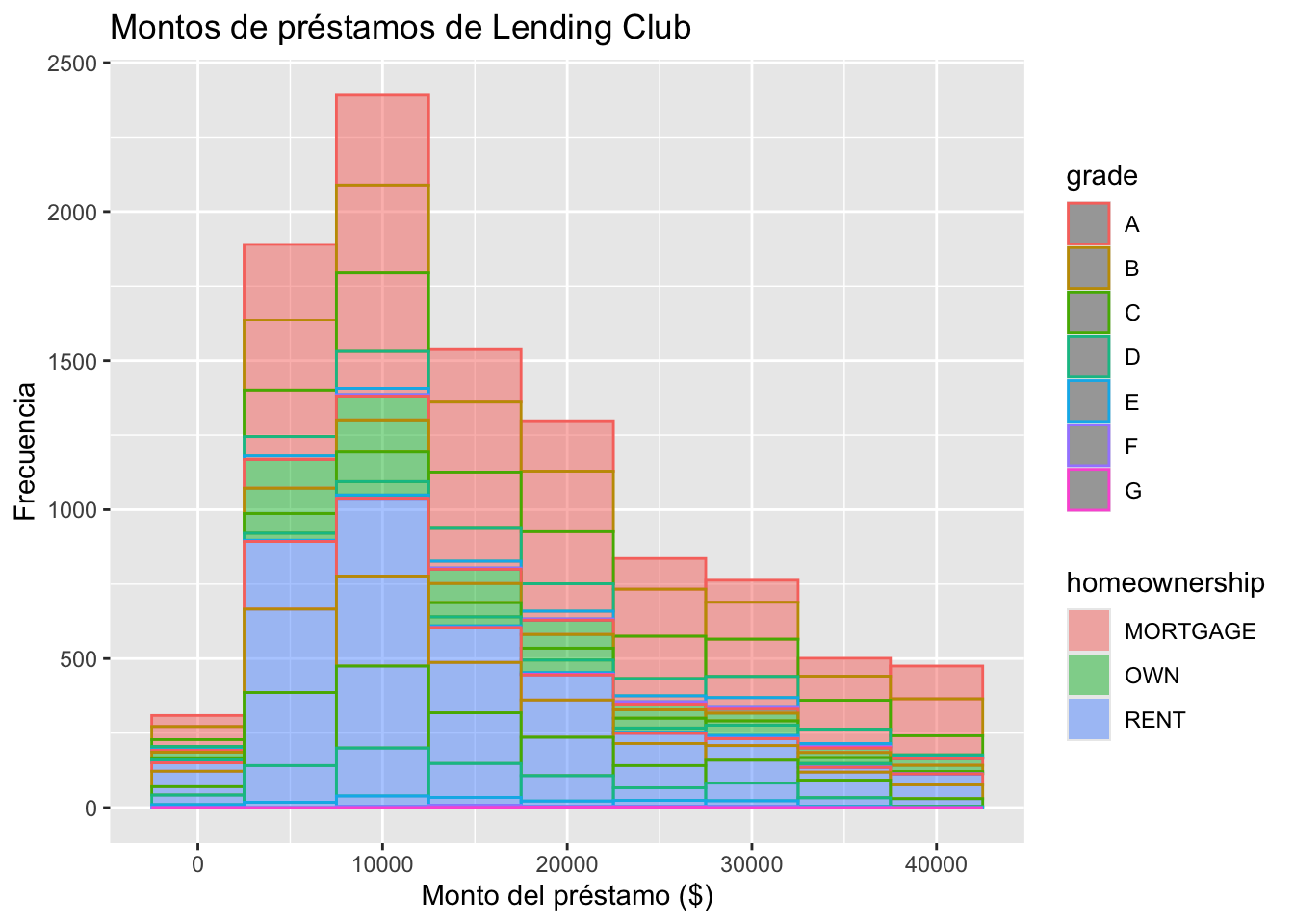
Agregar una variable categórica
ggplot(loans, aes(x = loan_amount, fill = homeownership, color = grade)) +
geom_histogram(binwidth = 5000, alpha = 0.5) +
labs(
x = "Monto del préstamo ($)",
y = "Frecuencia",
title = "Montos de préstamos de Lending Club"
)
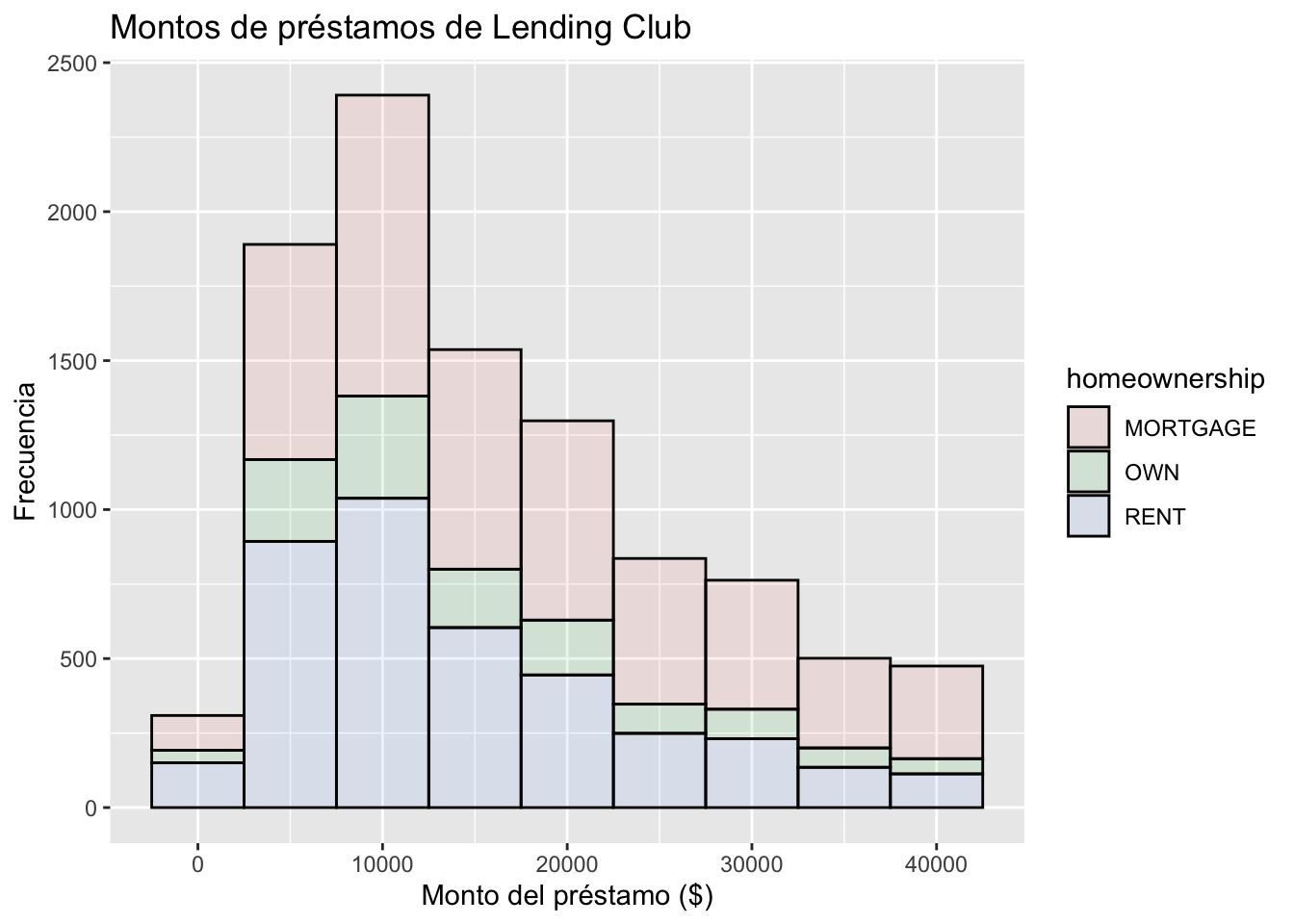
ggplot(loans, aes(x = loan_amount, fill = homeownership)) +
geom_histogram(binwidth = 5000, alpha = 0.1, color = "black") +
labs(
x = "Monto del préstamo ($)",
y = "Frecuencia",
title = "Montos de préstamos de Lending Club"
)
Facetar por variable categórica
ggplot(loans, aes(x = loan_amount, fill = homeownership)) +
geom_histogram(binwidth = 5000) +
facet_wrap(~homeownership, nrow = 3) +
labs(
x = "Monto del préstamo ($)",
y = "Frecuencia",
title = "Montos de préstamos de Lending Club",
fill = "Tipo de\nvivienda"
) +
cowplot::theme_minimal_grid() +
theme(legend.position = "none")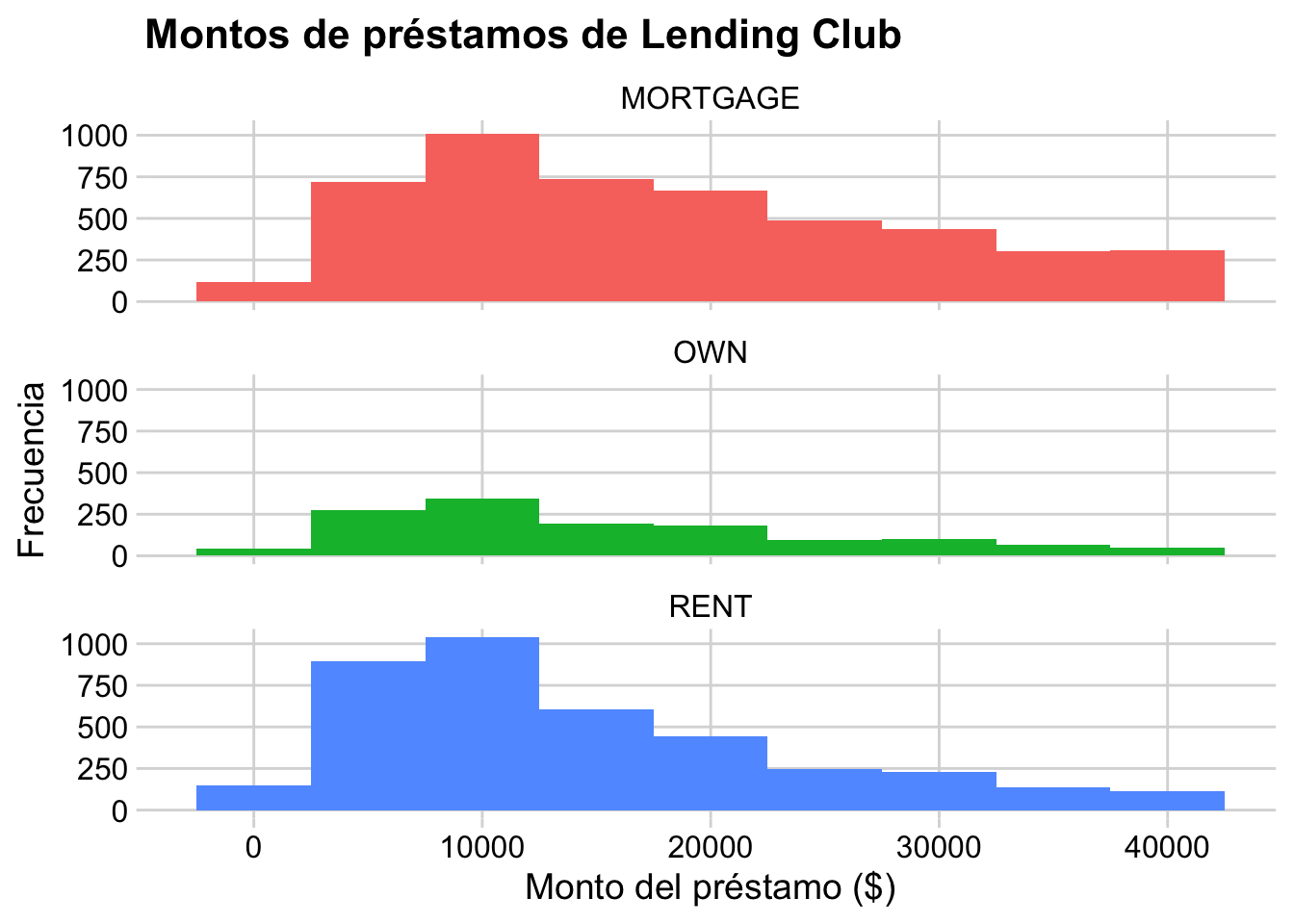
6.3.2 Gráfico de densidad
Gráfico de densidad básico
ggplot(loans, aes(x = loan_amount)) +
geom_density()
Ajustando el ancho de banda
ggplot(loans, aes(x = loan_amount)) +
geom_density(adjust = 0.5)
ggplot(loans, aes(x = loan_amount)) +
geom_density(adjust = 1)
ggplot(loans, aes(x = loan_amount)) +
geom_density(adjust = 2)
Personalización del gráfico de densidad
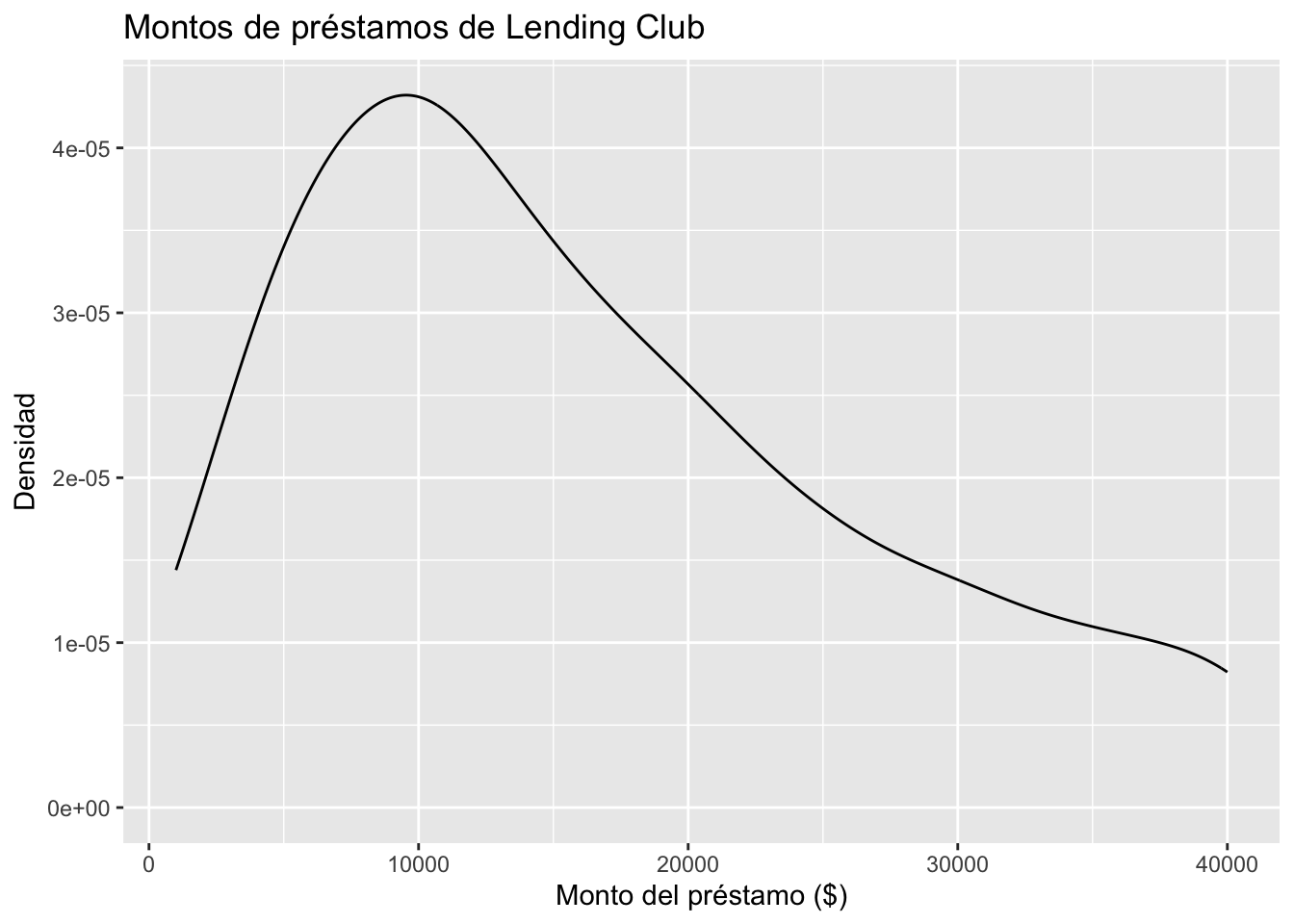
ggplot(loans, aes(x = loan_amount)) +
geom_density(adjust = 2) +
labs(
x = "Monto del préstamo ($)",
y = "Densidad",
title = "Montos de préstamos de Lending Club"
)
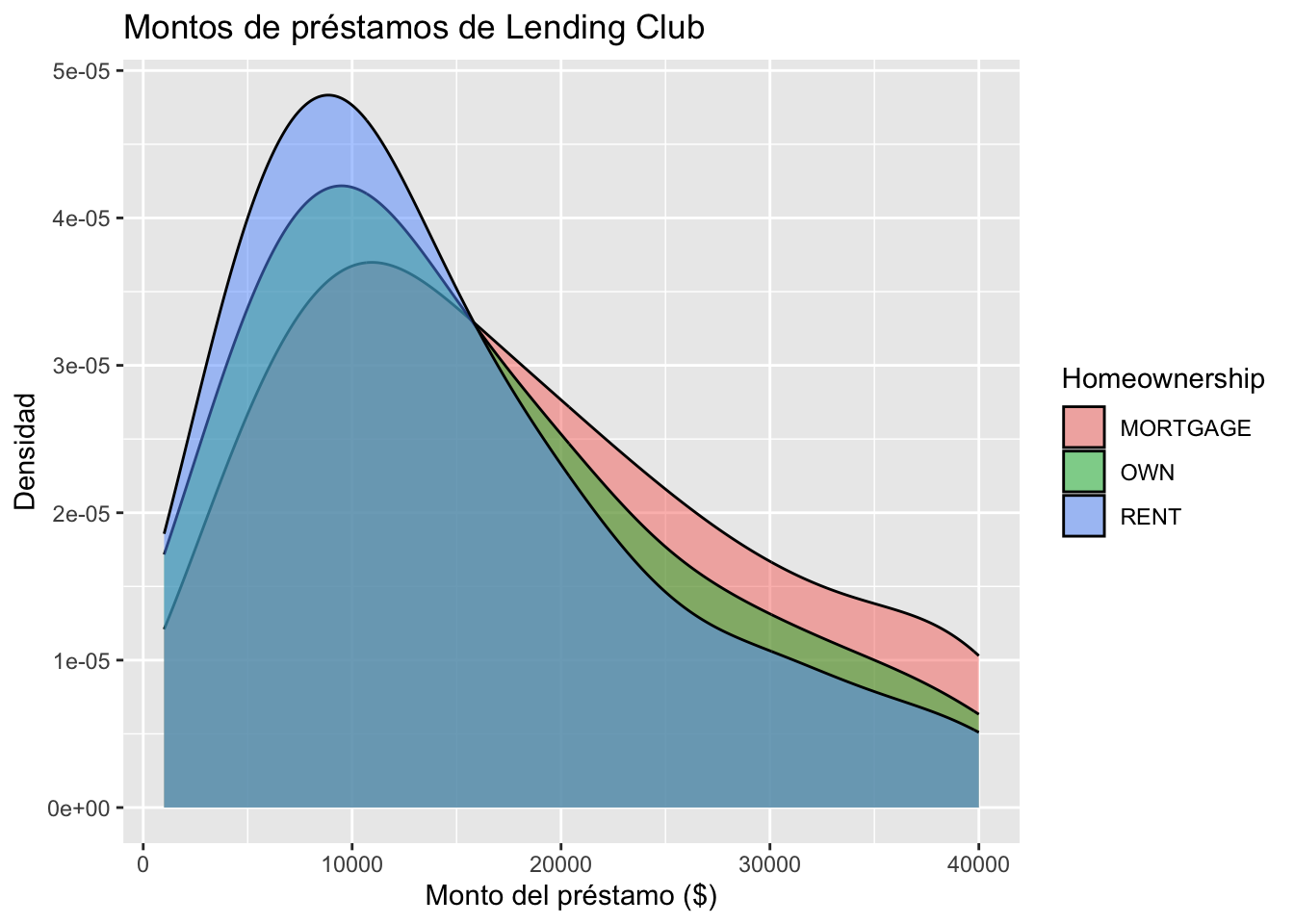
Agregar una variable categórica al gráfico de densidad
ggplot(loans, aes(x = loan_amount, fill = homeownership)) +
geom_density(adjust = 2, alpha = 0.5) +
labs(
x = "Monto del préstamo ($)",
y = "Densidad",
title = "Montos de préstamos de Lending Club",
fill = "Homeownership"
)
6.3.3 Gráfico de caja
Gráfico de caja básico
ggplot(loans, aes(x = interest_rate)) +
geom_boxplot()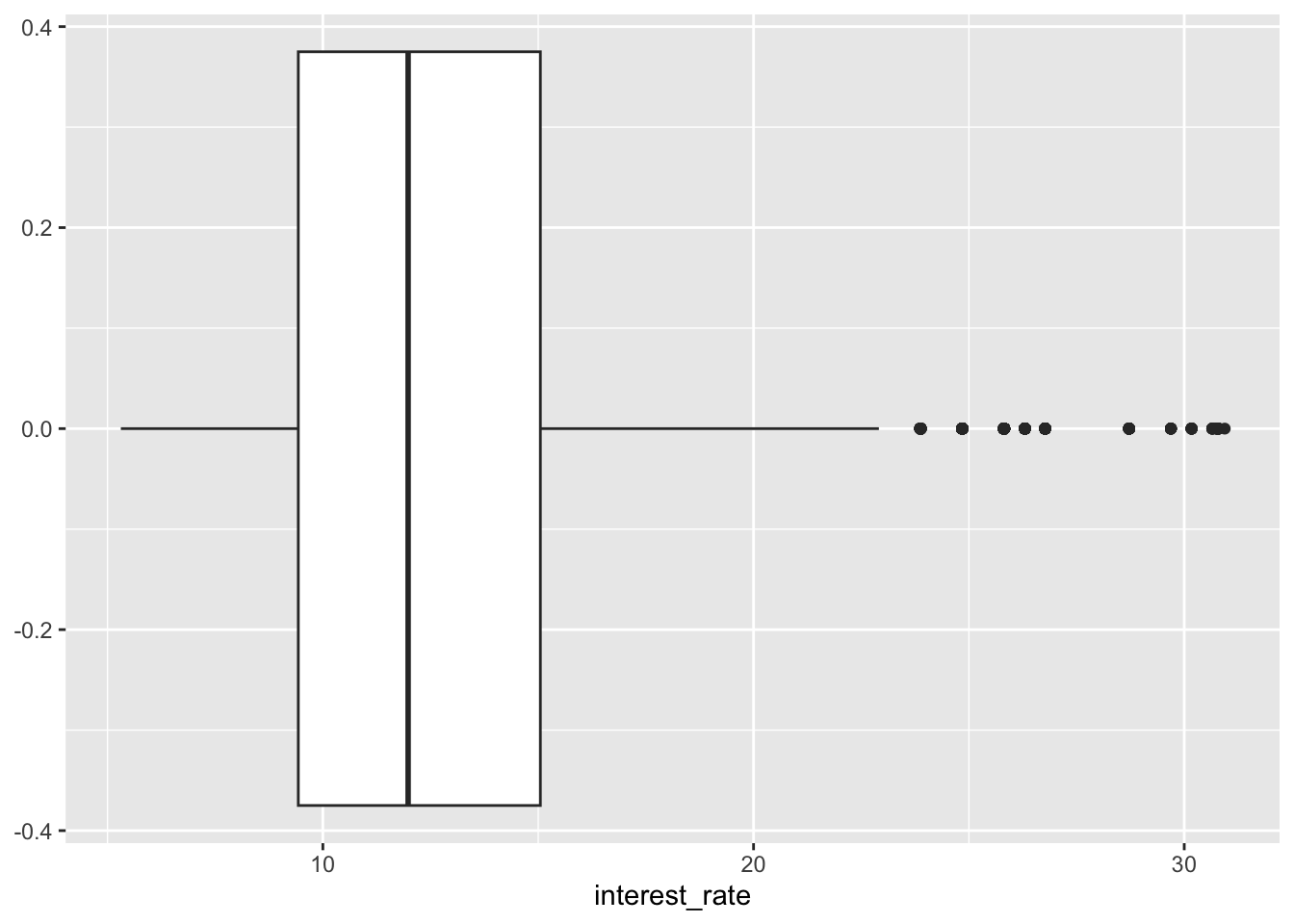
Gráfico de caja con valores atípicos
ggplot(loans, aes(x = annual_income)) +
geom_boxplot()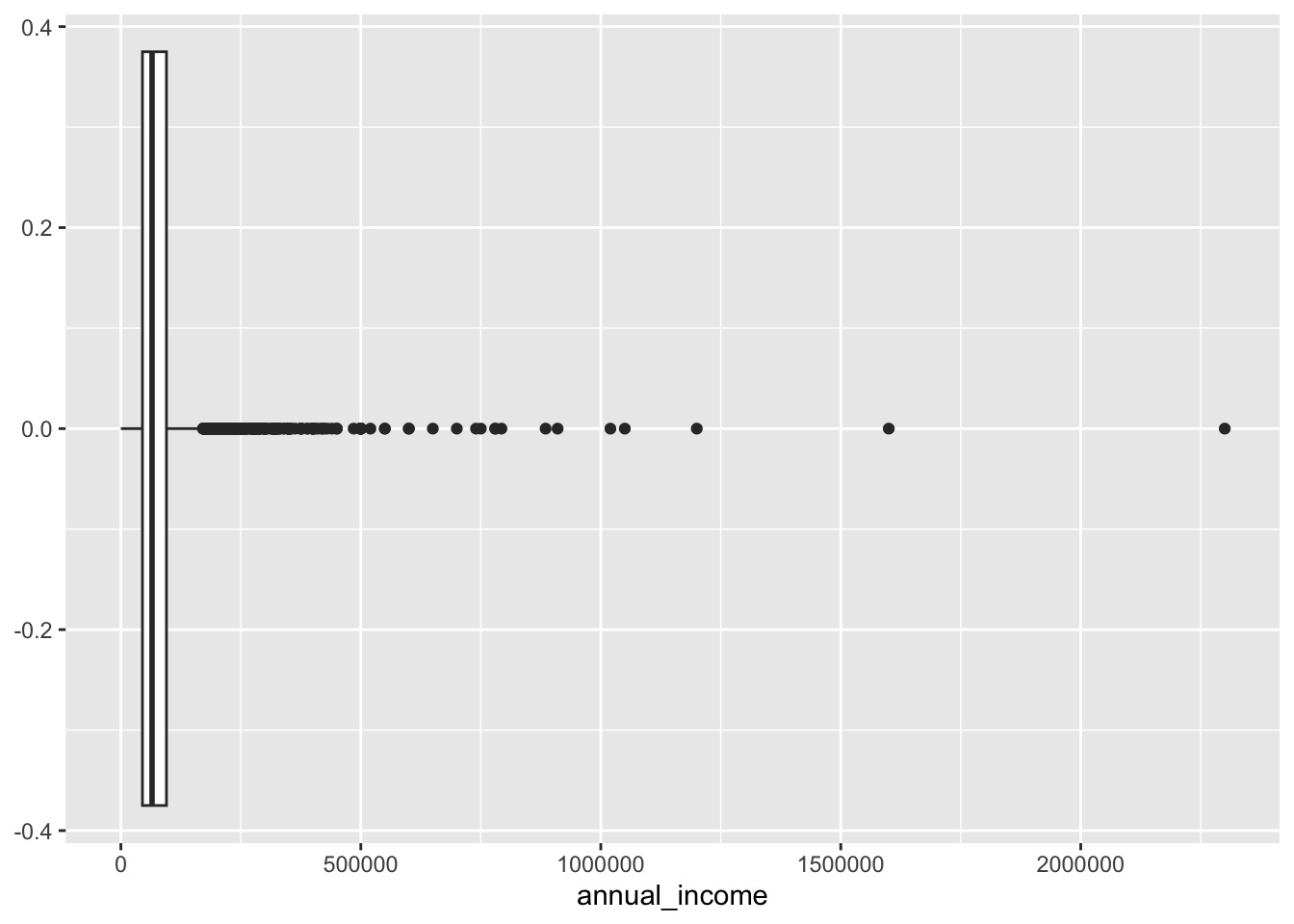
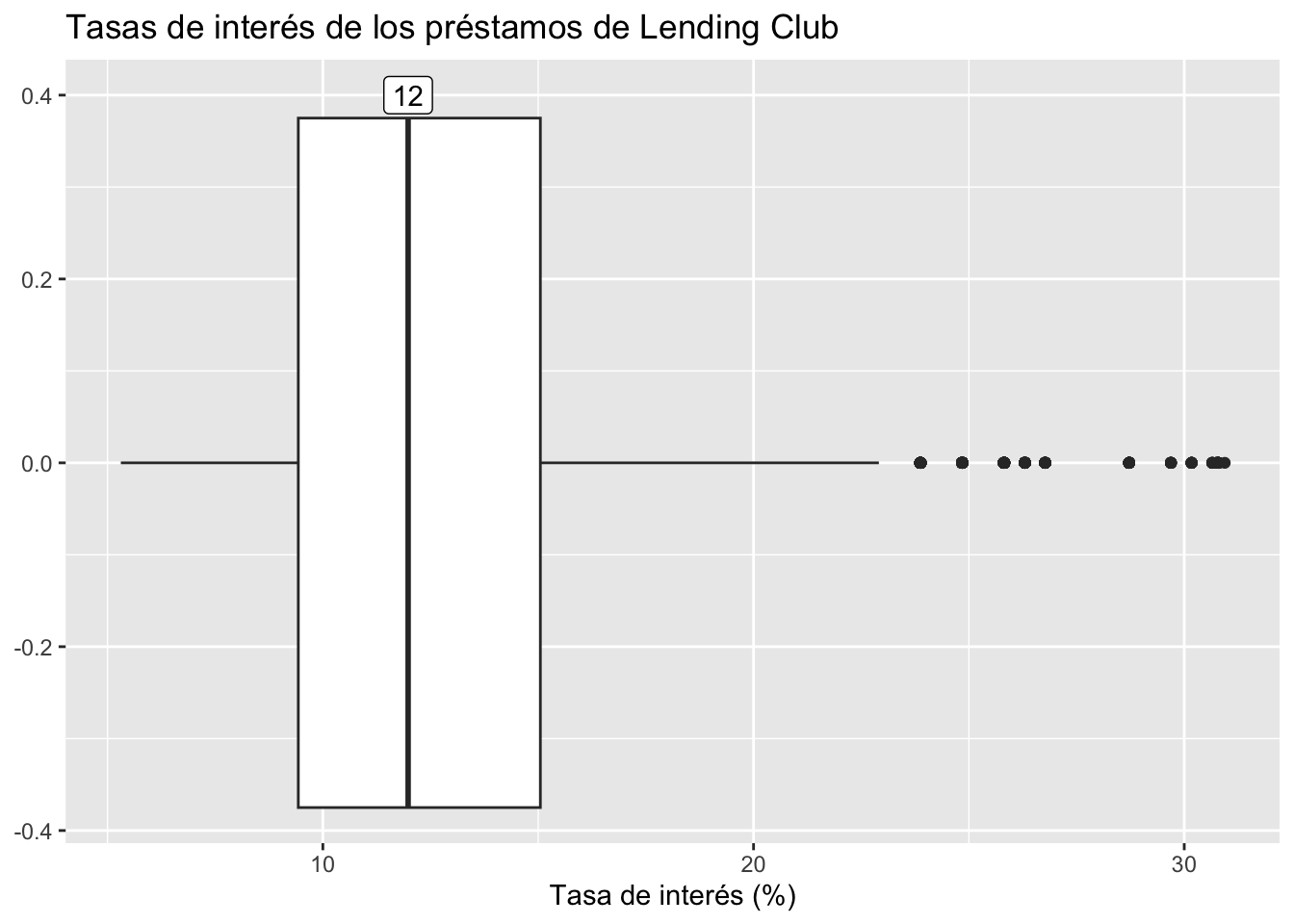
Personalización de gráficos de caja
valor_mediana <- median(loans$interest_rate)
ggplot(data = loans, mapping = aes(x = interest_rate)) +
geom_boxplot() +
labs(
x = "Tasa de interés (%)",
y = NULL,
title = "Tasas de interés de los préstamos de Lending Club"
) +
geom_label(
data = data.frame(
x = valor_mediana,
y = 0.4,
label = as.character(round(valor_mediana))
),
mapping = aes(x = x, y = y, label = label)
)
# theme(
# axis.ticks.y = element_blank(),
# axis.text.y = element_blank()
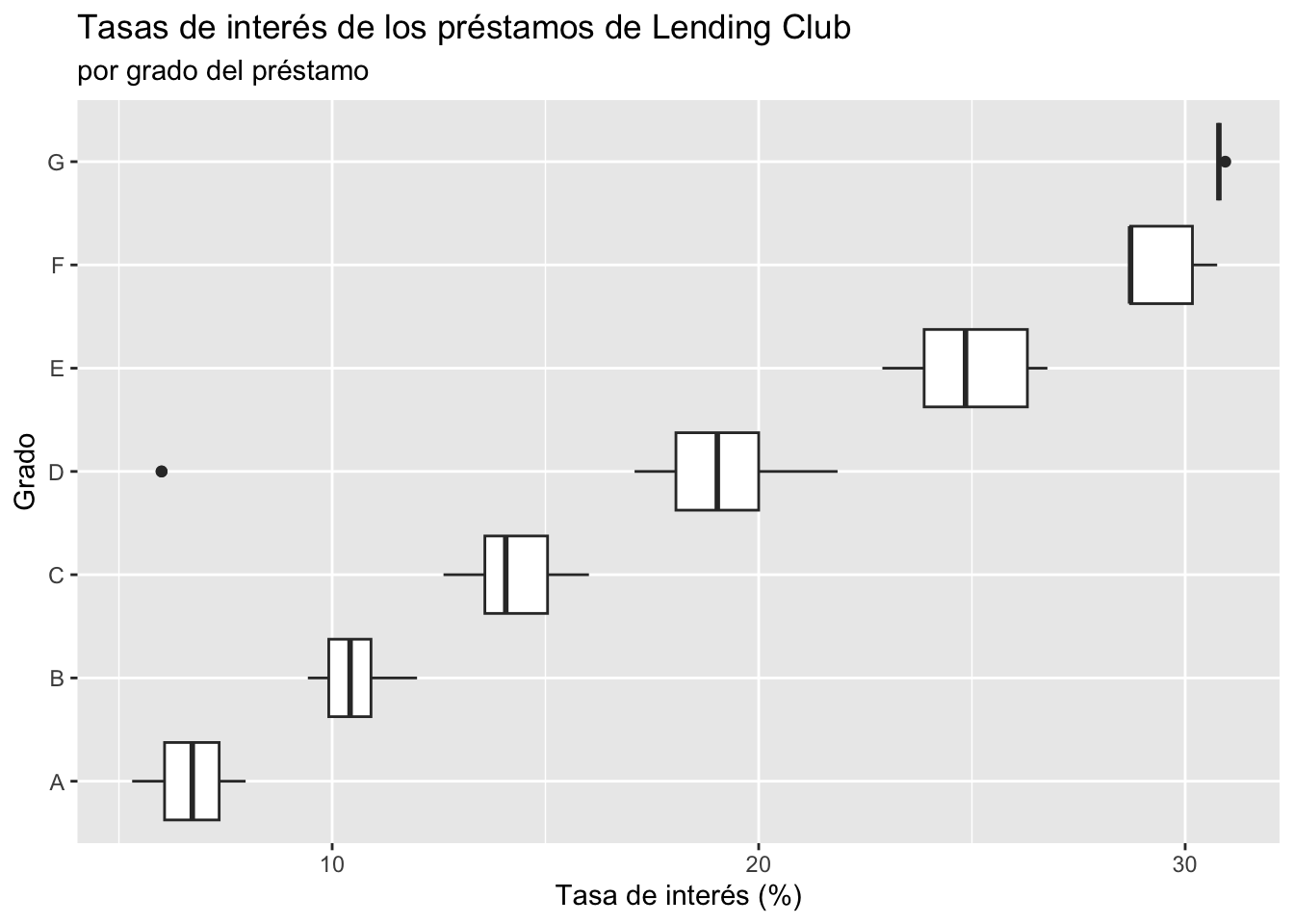
# )Agregar una variable categórica al gráfico de caja
ggplot(loans, aes(x = interest_rate, y = grade)) +
geom_boxplot() +
labs(
x = "Tasa de interés (%)",
y = "Grado",
title = "Tasas de interés de los préstamos de Lending Club",
subtitle = "por grado del préstamo"
)
6.3.4 Relación entre variables numéricas
6.3.4.1 Gráfico de dispersión
ggplot(loans, aes(x = debt_to_income, y = interest_rate)) +
geom_point()Warning: Removed 24 rows containing missing values or values outside the scale range
(`geom_point()`).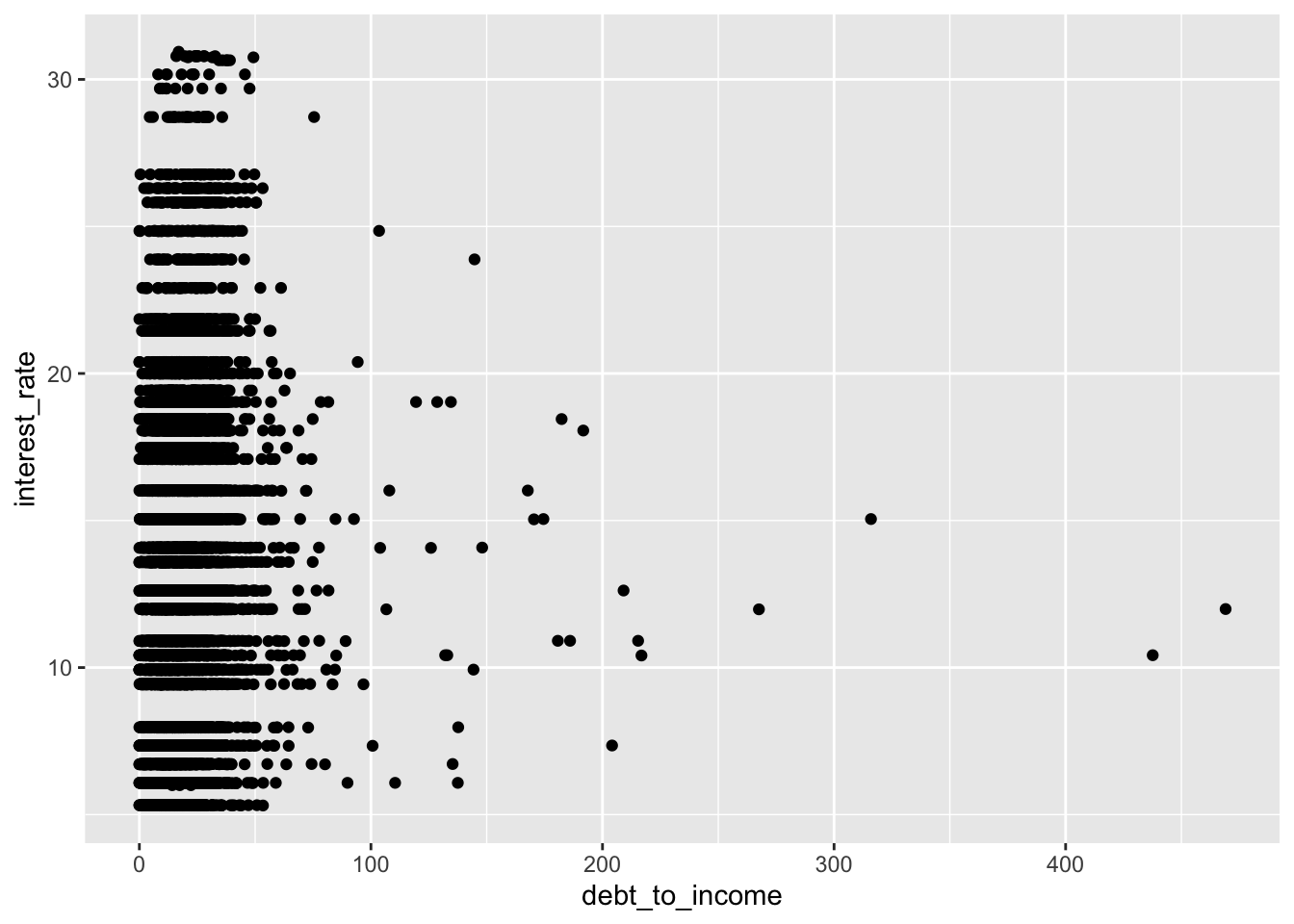
6.3.4.2 Gráfico hexagonal
ggplot(loans, aes(x = debt_to_income, y = interest_rate)) +
geom_hex()Warning: Removed 24 rows containing non-finite outside the scale range
(`stat_binhex()`).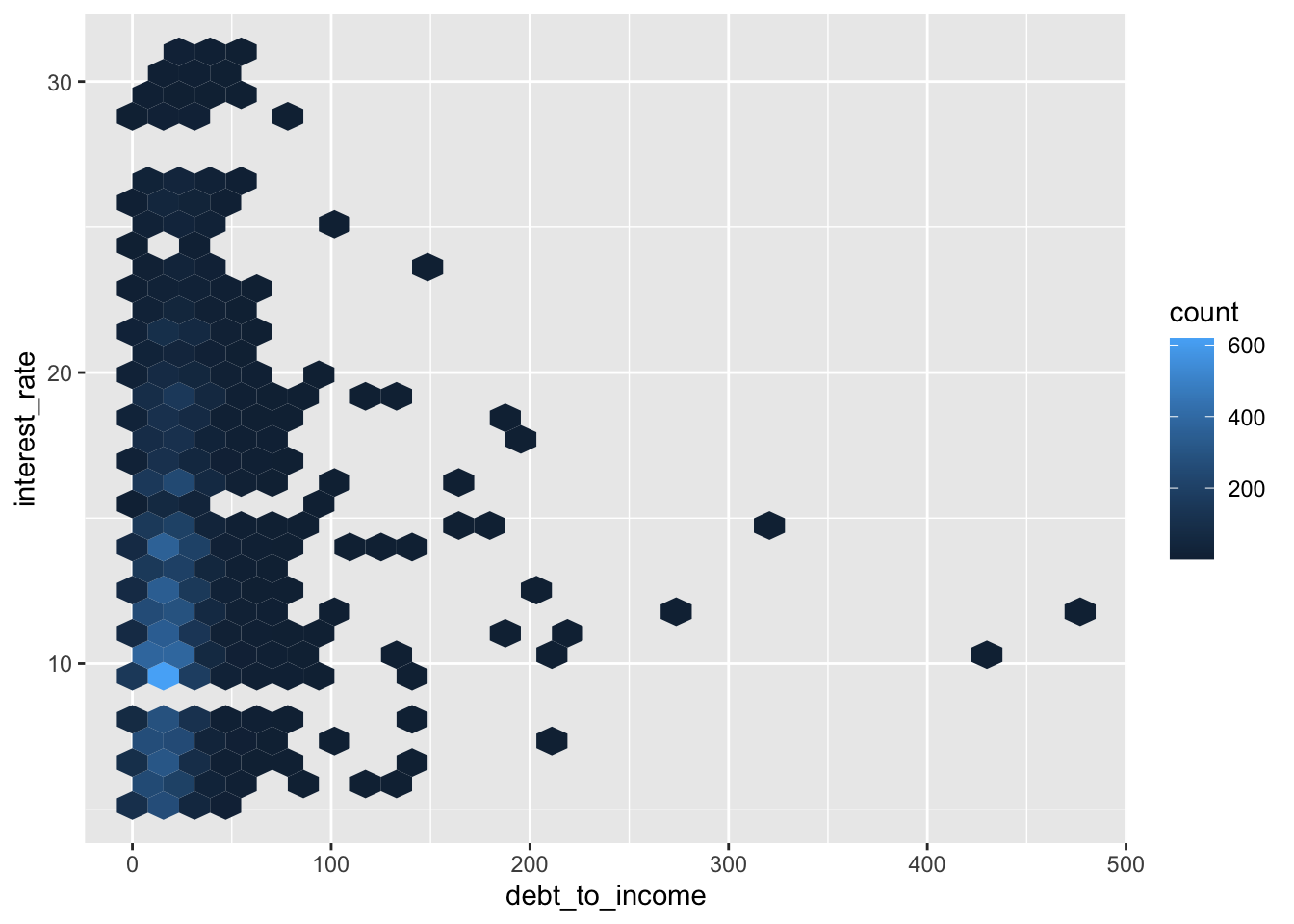
Gráfico hexagonal con filtro
ggplot(
loans %>% filter(debt_to_income < 100),
aes(x = debt_to_income, y = interest_rate)
) +
geom_hex()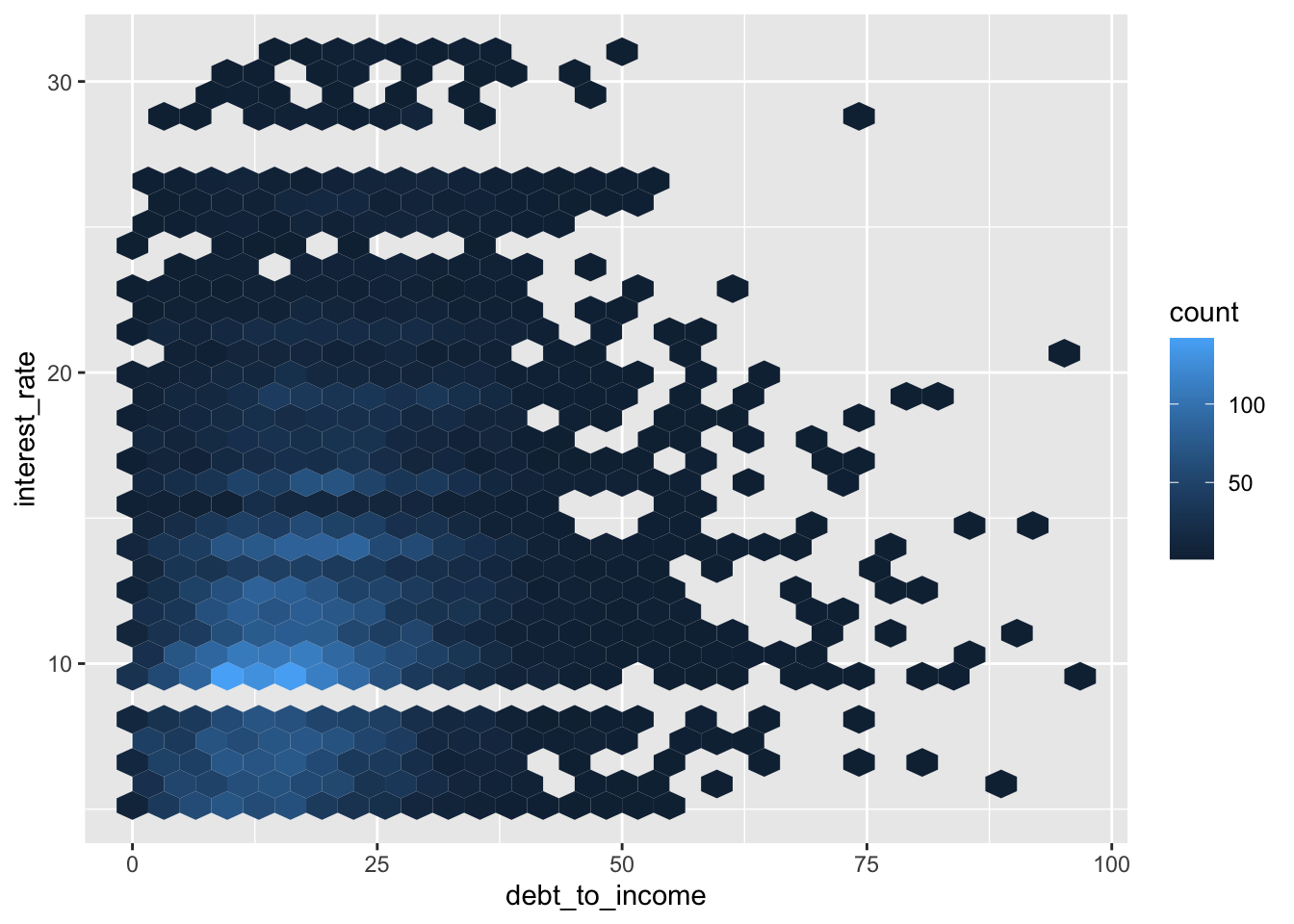
6.3.4.3 Gráfico de barras
Gráfico de barras básico
ggplot(loans, aes(x = homeownership)) +
geom_bar()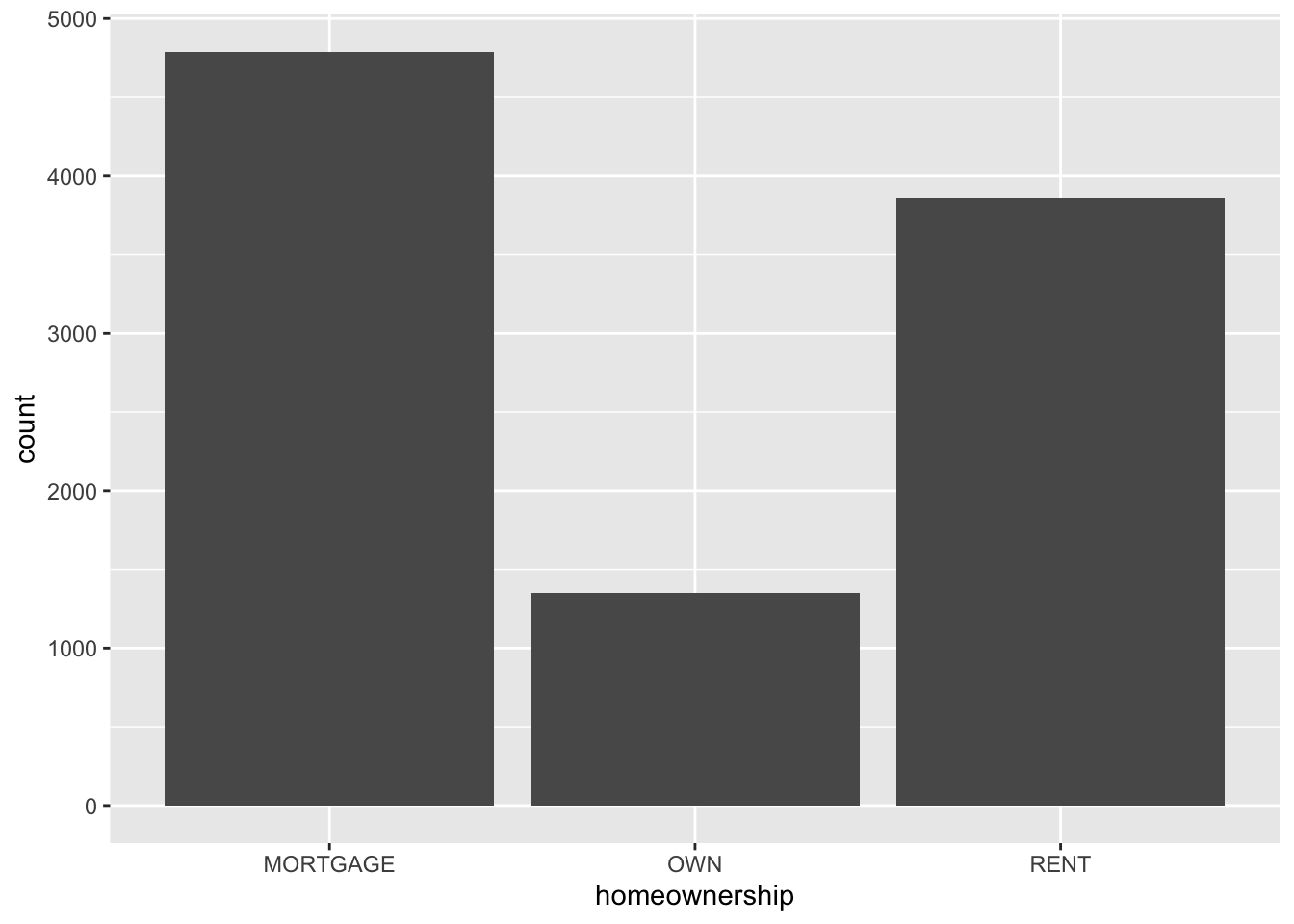
Gráfico de barras segmentado
ggplot(loans, aes(x = homeownership, fill = grade)) +
geom_bar()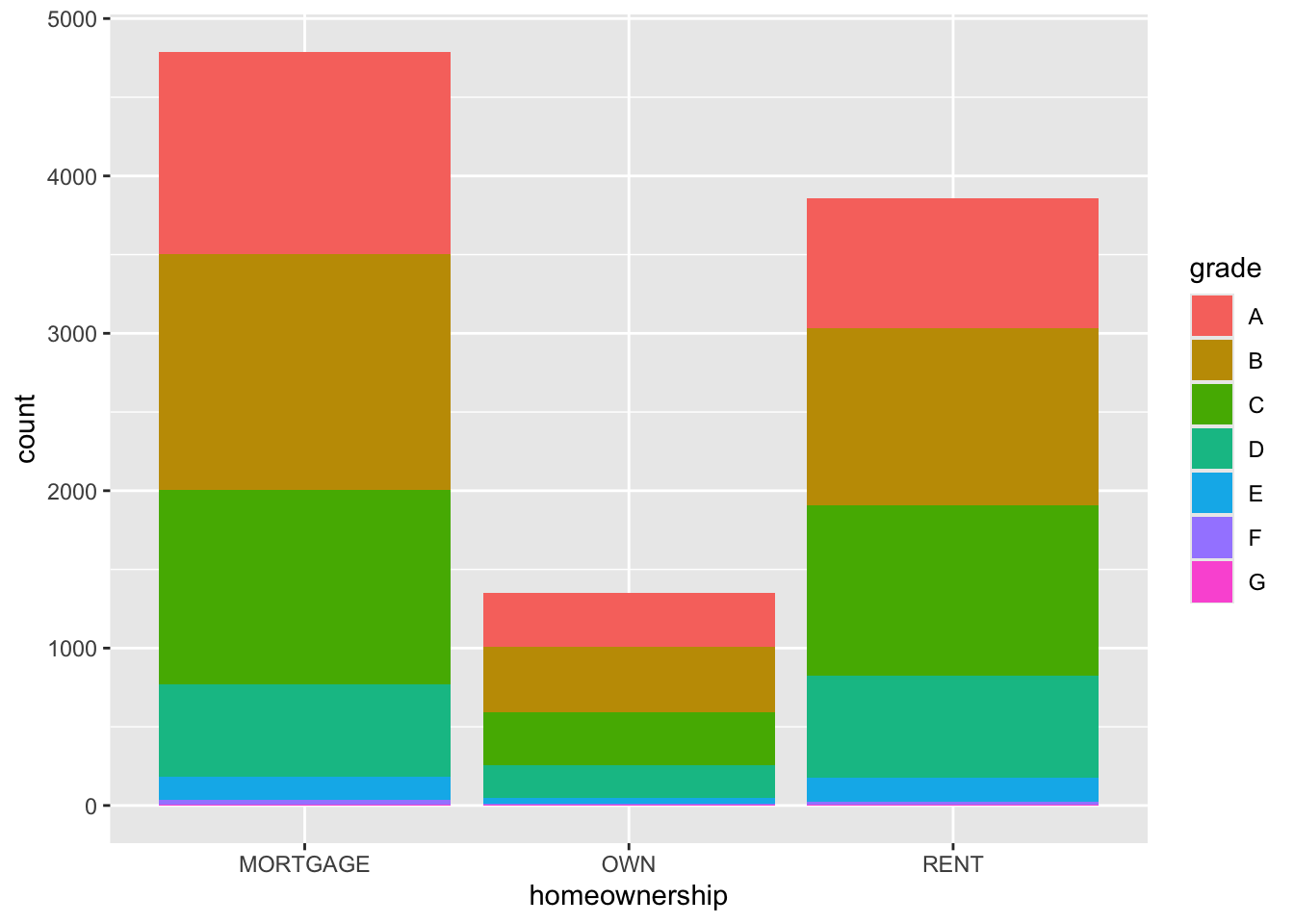
Gráfico de barras segmentado por proporción
ggplot(loans, aes(x = homeownership, fill = grade)) +
geom_bar(position = "fill")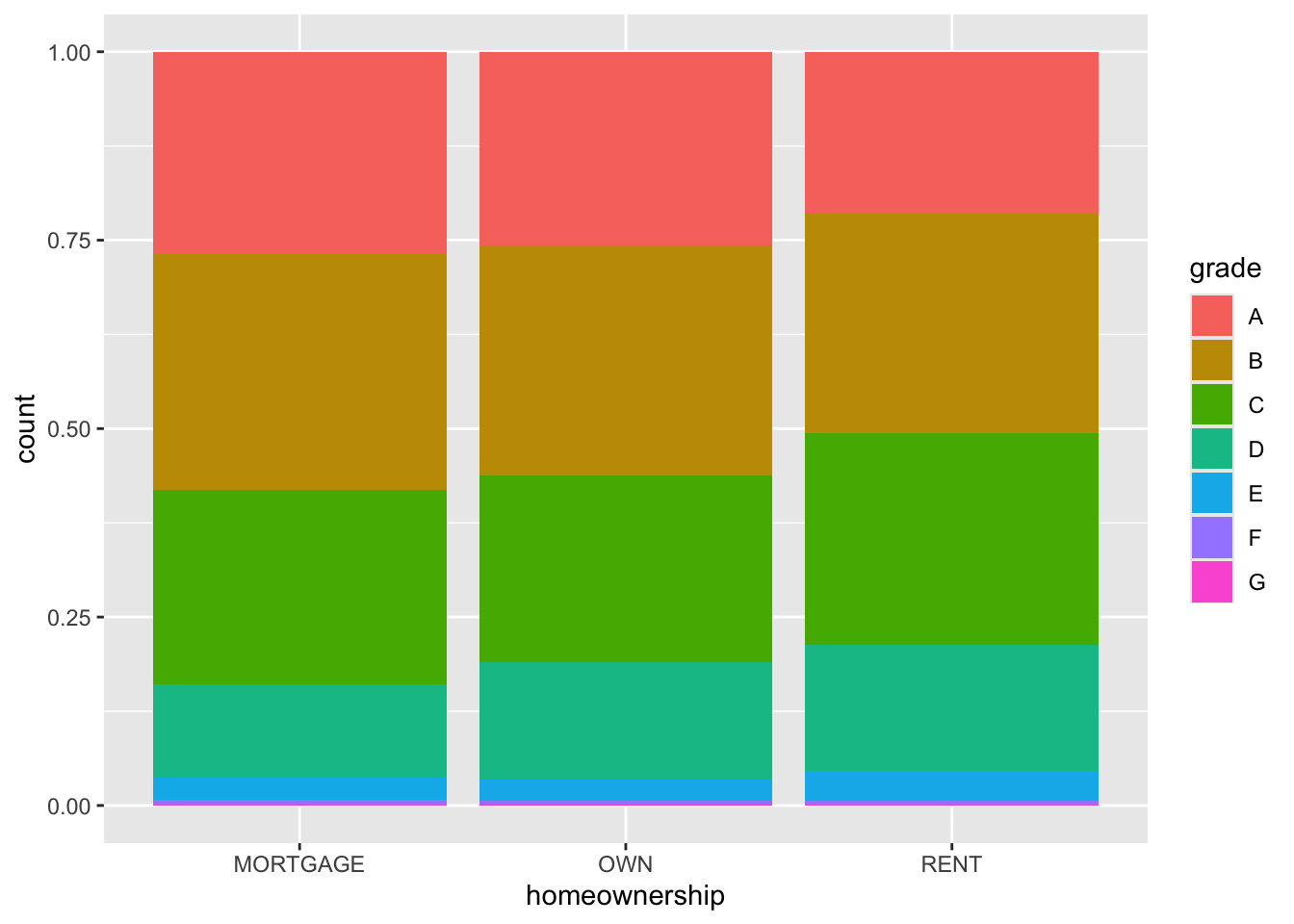
Personalización de gráficos de barras
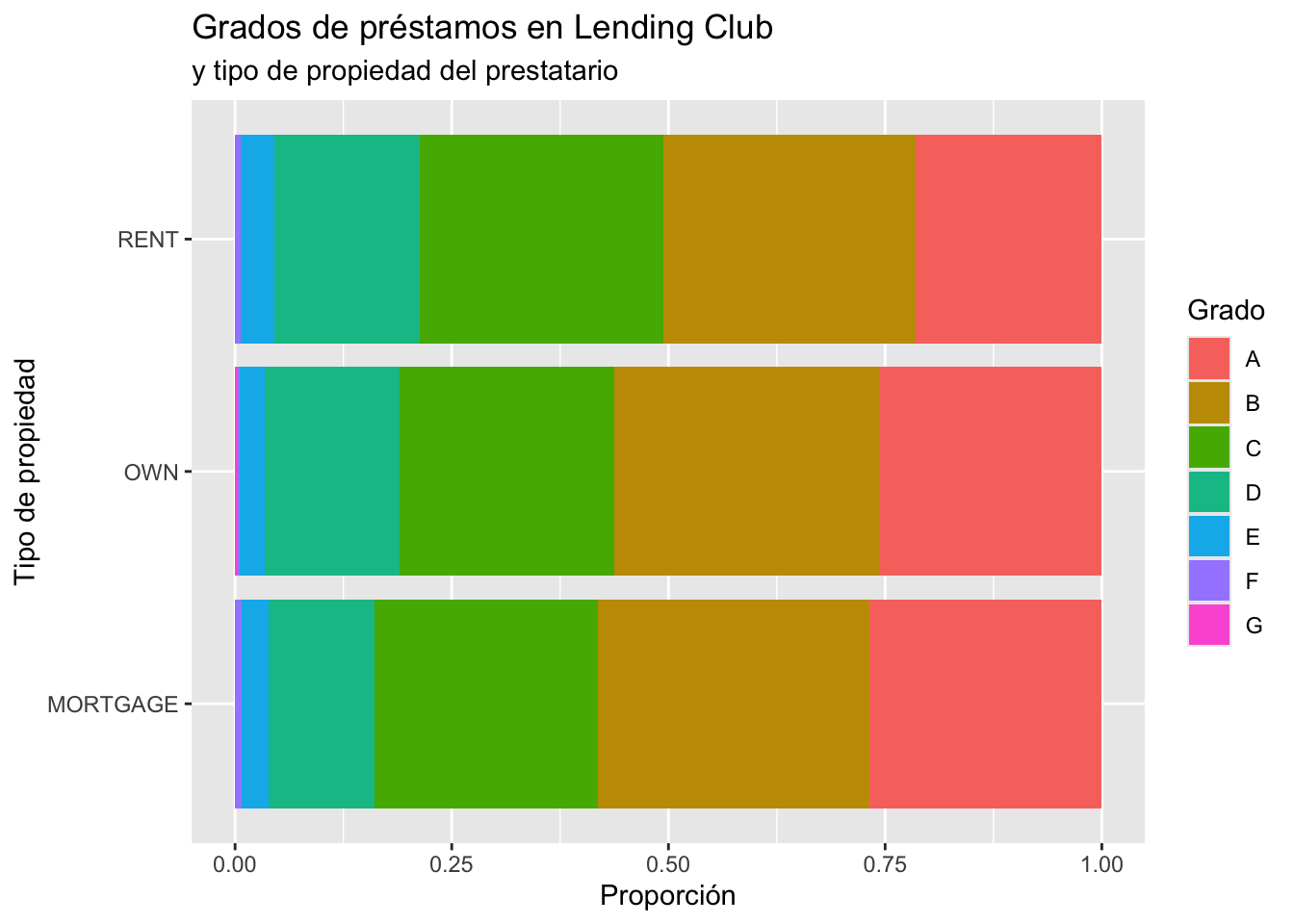
ggplot(loans, aes(y = homeownership, fill = grade)) +
geom_bar(position = "fill") +
labs(
x = "Proporción",
y = "Tipo de propiedad",
fill = "Grado",
title = "Grados de préstamos en Lending Club",
subtitle = "y tipo de propiedad del prestatario"
)
6.3.5 Relaciones entre variables numéricas y categóricas
Violin plots
ggplot(loans, aes(x = homeownership, y = loan_amount)) +
geom_violin()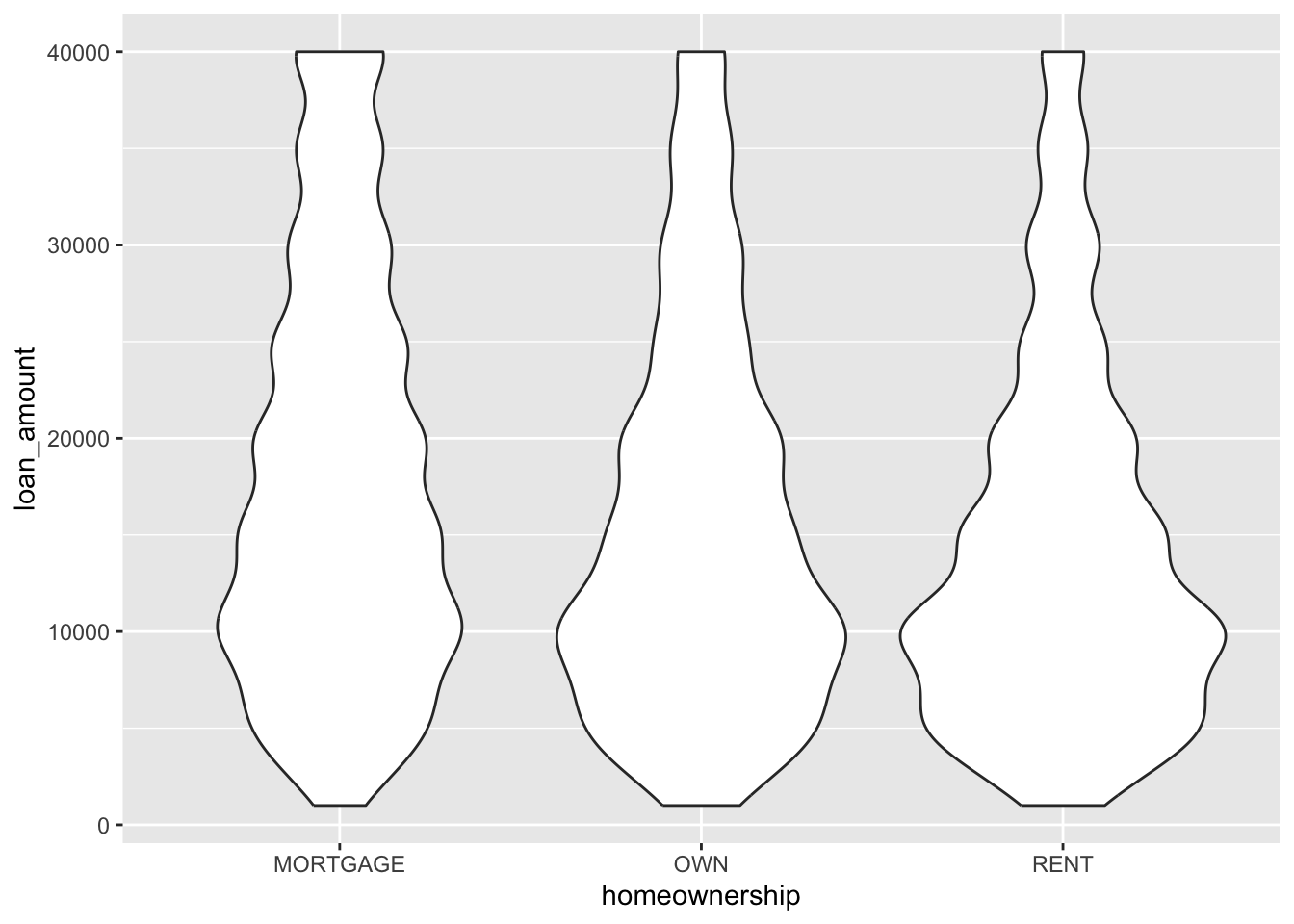
Ridge plots
library(ggridges)
ggplot(loans, aes(x = loan_amount, y = grade, fill = grade, color = grade)) +
geom_density_ridges(alpha = 0.5)Picking joint bandwidth of 2360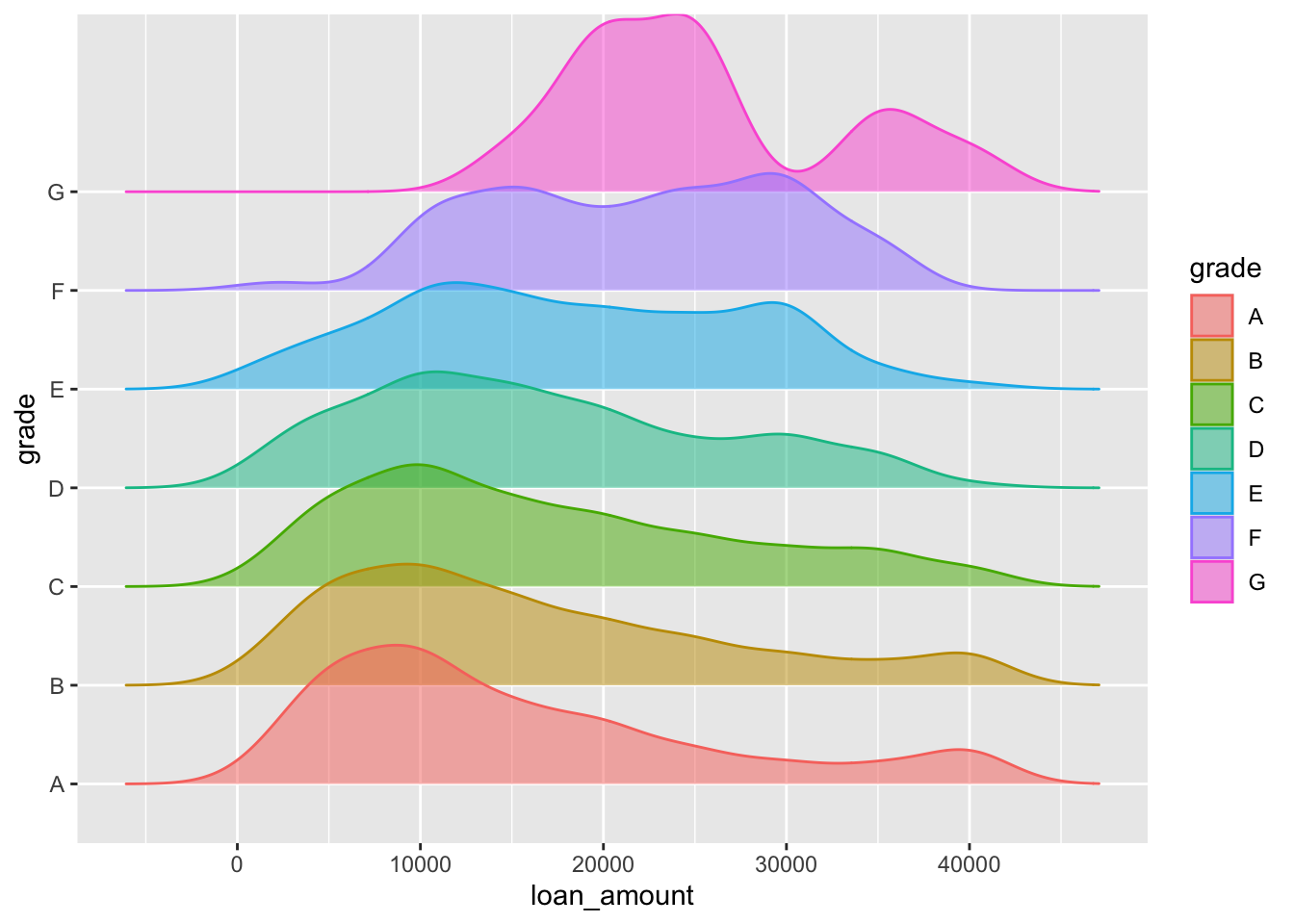
6.4 Manipulación de datos con dplyr
Ahora que hemos visualizado los datos, vamos a realizar algunas operaciones comunes de manipulación utilizando dplyr.
6.4.1 Seleccionar columnas
Vamos a seleccionar algunas columnas clave de nuestro dataset: país, continente y esperanza de vida.
## Seleccionar columnas con dplyr
(gapminder_selected <- gapminder %>%
select(country, continent, lifeExp))# A tibble: 1,704 × 3
country continent lifeExp
<chr> <chr> <dbl>
1 Afghanistan Asia 28.8
2 Afghanistan Asia 30.3
3 Afghanistan Asia 32.0
4 Afghanistan Asia 34.0
5 Afghanistan Asia 36.1
6 Afghanistan Asia 38.4
7 Afghanistan Asia 39.9
8 Afghanistan Asia 40.8
9 Afghanistan Asia 41.7
10 Afghanistan Asia 41.8
# ℹ 1,694 more rows## Comparación con base R
(gapminder_base_selected <- gapminder[, c("country", "continent", "lifeExp")])# A tibble: 1,704 × 3
country continent lifeExp
<chr> <chr> <dbl>
1 Afghanistan Asia 28.8
2 Afghanistan Asia 30.3
3 Afghanistan Asia 32.0
4 Afghanistan Asia 34.0
5 Afghanistan Asia 36.1
6 Afghanistan Asia 38.4
7 Afghanistan Asia 39.9
8 Afghanistan Asia 40.8
9 Afghanistan Asia 41.7
10 Afghanistan Asia 41.8
# ℹ 1,694 more rows6.4.2 Filtrar observaciones
Vamos a filtrar las observaciones correspondientes al año 1957.
# Filtrar datos de 1957 con dplyr
(gapminder_1957 <- gapminder %>%
filter(year == 1957))# A tibble: 142 × 6
country year pop continent lifeExp gdpPercap
<chr> <dbl> <dbl> <chr> <dbl> <dbl>
1 Afghanistan 1957 9240934 Asia 30.3 821.
2 Albania 1957 1476505 Europe 59.3 1942.
3 Algeria 1957 10270856 Africa 45.7 3014.
4 Angola 1957 4561361 Africa 32.0 3828.
5 Argentina 1957 19610538 Americas 64.4 6857.
6 Australia 1957 9712569 Oceania 70.3 10950.
7 Austria 1957 6965860 Europe 67.5 8843.
8 Bahrain 1957 138655 Asia 53.8 11636.
9 Bangladesh 1957 51365468 Asia 39.3 662.
10 Belgium 1957 8989111 Europe 69.2 9715.
# ℹ 132 more rows# Comparación con base R
(gapminder_base_1957 <- gapminder[gapminder$year == 1957, ])# A tibble: 142 × 6
country year pop continent lifeExp gdpPercap
<chr> <dbl> <dbl> <chr> <dbl> <dbl>
1 Afghanistan 1957 9240934 Asia 30.3 821.
2 Albania 1957 1476505 Europe 59.3 1942.
3 Algeria 1957 10270856 Africa 45.7 3014.
4 Angola 1957 4561361 Africa 32.0 3828.
5 Argentina 1957 19610538 Americas 64.4 6857.
6 Australia 1957 9712569 Oceania 70.3 10950.
7 Austria 1957 6965860 Europe 67.5 8843.
8 Bahrain 1957 138655 Asia 53.8 11636.
9 Bangladesh 1957 51365468 Asia 39.3 662.
10 Belgium 1957 8989111 Europe 69.2 9715.
# ℹ 132 more rows6.4.3 Crear nuevas columnas con mutate
Vamos a crear una nueva columna que exprese el PIB per cápita en miles de dólares.
# Crear nueva columna con mutate
(gapminder <- gapminder %>%
mutate(gdpPercap_thousands = gdpPercap / 1000))# A tibble: 1,704 × 7
country year pop continent lifeExp gdpPercap gdpPercap_thousands
<chr> <dbl> <dbl> <chr> <dbl> <dbl> <dbl>
1 Afghanistan 1952 8425333 Asia 28.8 779. 0.779
2 Afghanistan 1957 9240934 Asia 30.3 821. 0.821
3 Afghanistan 1962 10267083 Asia 32.0 853. 0.853
4 Afghanistan 1967 11537966 Asia 34.0 836. 0.836
5 Afghanistan 1972 13079460 Asia 36.1 740. 0.740
6 Afghanistan 1977 14880372 Asia 38.4 786. 0.786
7 Afghanistan 1982 12881816 Asia 39.9 978. 0.978
8 Afghanistan 1987 13867957 Asia 40.8 852. 0.852
9 Afghanistan 1992 16317921 Asia 41.7 649. 0.649
10 Afghanistan 1997 22227415 Asia 41.8 635. 0.635
# ℹ 1,694 more rows# Comparación con base R
gapminder$gdpPercap_thousands <- gapminder$gdpPercap / 10006.4.4 Resumir datos con summarise
Vamos a calcular la esperanza de vida promedio por continente.
# Resumir datos con dplyr
(gapminder_summary <- gapminder %>%
group_by(continent) %>%
summarise(avg_lifeExp = mean(lifeExp)))# A tibble: 5 × 2
continent avg_lifeExp
<chr> <dbl>
1 Africa 48.9
2 Americas 64.7
3 Asia 60.1
4 Europe 71.9
5 Oceania 74.3# Comparación con base R
(gapminder_base_summary <-
aggregate(lifeExp ~ continent, data = gapminder, FUN = mean)) continent lifeExp
1 Africa 48.86533
2 Americas 64.65874
3 Asia 60.06490
4 Europe 71.90369
5 Oceania 74.326216.4.5 Ordenar datos con arrange
Finalmente, vamos a ordenar los datos según la esperanza de vida de forma descendente.
# Ordenar datos con dplyr
(gapminder_sorted <- gapminder %>%
arrange(desc(lifeExp)))# A tibble: 1,704 × 7
country year pop continent lifeExp gdpPercap gdpPercap_thousands
<chr> <dbl> <dbl> <chr> <dbl> <dbl> <dbl>
1 Japan 2007 1.27e8 Asia 82.6 31656. 31.7
2 Hong Kong China 2007 6.98e6 Asia 82.2 39725. 39.7
3 Japan 2002 1.27e8 Asia 82 28605. 28.6
4 Iceland 2007 3.02e5 Europe 81.8 36181. 36.2
5 Switzerland 2007 7.55e6 Europe 81.7 37506. 37.5
6 Hong Kong China 2002 6.76e6 Asia 81.5 30209. 30.2
7 Australia 2007 2.04e7 Oceania 81.2 34435. 34.4
8 Spain 2007 4.04e7 Europe 80.9 28821. 28.8
9 Sweden 2007 9.03e6 Europe 80.9 33860. 33.9
10 Israel 2007 6.43e6 Asia 80.7 25523. 25.5
# ℹ 1,694 more rows# Comparación con base R
gapminder_base_sorted <- gapminder[order(-gapminder$lifeExp), ]6.4.6 Condicionales y bucles avanzados
6.4.6.1 Condicionales
Usar if() para verificar si existen registros del año 2002 en el dataset.
# Verificar si hay datos del año 2002
if (any(gapminder$year == 2002)) {
print("Se encontraron registros para el año 2002.")
} else {
print("No se encontraron registros para el año 2002.")
}[1] "Se encontraron registros para el año 2002."(gapminder |>
mutate(registros_2022 = case_when(
year == 2022 ~ TRUE,
TRUE ~ FALSE
)))# A tibble: 1,704 × 8
country year pop continent lifeExp gdpPercap gdpPercap_thousands
<chr> <dbl> <dbl> <chr> <dbl> <dbl> <dbl>
1 Afghanistan 1952 8425333 Asia 28.8 779. 0.779
2 Afghanistan 1957 9240934 Asia 30.3 821. 0.821
3 Afghanistan 1962 10267083 Asia 32.0 853. 0.853
4 Afghanistan 1967 11537966 Asia 34.0 836. 0.836
5 Afghanistan 1972 13079460 Asia 36.1 740. 0.740
6 Afghanistan 1977 14880372 Asia 38.4 786. 0.786
7 Afghanistan 1982 12881816 Asia 39.9 978. 0.978
8 Afghanistan 1987 13867957 Asia 40.8 852. 0.852
9 Afghanistan 1992 16317921 Asia 41.7 649. 0.649
10 Afghanistan 1997 22227415 Asia 41.8 635. 0.635
# ℹ 1,694 more rows
# ℹ 1 more variable: registros_2022 <lgl>6.4.6.2 Bucles
Recorrer los datos por continente e imprimir si la esperanza de vida media es menor o mayor de 50 años.
# Bucles en base R
for (iContinent in unique(gapminder$continent)) {
avg_lifeExp <- mean(gapminder %>%
filter(continent == iContinent) %>%
pull(lifeExp))
cat("Esperanza de vida promedio en", iContinent, "es", avg_lifeExp, "\n")
}Esperanza de vida promedio en Asia es 60.0649
Esperanza de vida promedio en Europe es 71.90369
Esperanza de vida promedio en Africa es 48.86533
Esperanza de vida promedio en Americas es 64.65874
Esperanza de vida promedio en Oceania es 74.32621 # Usar tapply para calcular la media de lifeExp por continente
tapply(X = gapminder$lifeExp, INDEX = gapminder$continent, FUN = mean) Africa Americas Asia Europe Oceania
48.86533 64.65874 60.06490 71.90369 74.32621 gapminder |>
group_by(continent) |>
nest() |>
mutate(avg_lifeExp = map(.x = data, .f = ~ mean(.x$lifeExp))) |>
select(continent, avg_lifeExp) |>
unnest(cols = c(avg_lifeExp))# A tibble: 5 × 2
# Groups: continent [5]
continent avg_lifeExp
<chr> <dbl>
1 Asia 60.1
2 Europe 71.9
3 Africa 48.9
4 Americas 64.7
5 Oceania 74.3title: “Trabajando con múltiples data frames” subtitle: “Data Science en una caja” author: “datasciencebox.org”
library(tidyverse)
library(knitr)
options(
dplyr.print_min = 10,
dplyr.print_max = 10
)6.5 Manejo de múltiples data frames
La información incluye a 10 mujeres en la ciencia que cambiaron el mundo.
professions <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d08-multi-df/data/scientists/professions.csv")Rows: 10 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (2): name, profession
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.dates <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d08-multi-df/data/scientists/dates.csv")Rows: 8 Columns: 3
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): name
dbl (2): birth_year, death_year
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.works <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d08-multi-df/data/scientists/works.csv")Rows: 9 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (2): name, known_for
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.professions# A tibble: 10 × 2
name profession
<chr> <chr>
1 Ada Lovelace Mathematician
2 Marie Curie Physicist and Chemist
3 Janaki Ammal Botanist
4 Chien-Shiung Wu Physicist
5 Katherine Johnson Mathematician
6 Rosalind Franklin Chemist
7 Vera Rubin Astronomer
8 Gladys West Mathematician
9 Flossie Wong-Staal Virologist and Molecular Biologist
10 Jennifer Doudna Biochemist dates# A tibble: 8 × 3
name birth_year death_year
<chr> <dbl> <dbl>
1 Janaki Ammal 1897 1984
2 Chien-Shiung Wu 1912 1997
3 Katherine Johnson 1918 2020
4 Rosalind Franklin 1920 1958
5 Vera Rubin 1928 2016
6 Gladys West 1930 NA
7 Flossie Wong-Staal 1947 NA
8 Jennifer Doudna 1964 NAworks# A tibble: 9 × 2
name known_for
<chr> <chr>
1 Ada Lovelace first computer algorithm
2 Marie Curie theory of radioactivity, discovery of elements polonium a…
3 Janaki Ammal hybrid species, biodiversity protection
4 Chien-Shiung Wu confim and refine theory of radioactive beta decy, Wu expe…
5 Katherine Johnson calculations of orbital mechanics critical to sending the …
6 Vera Rubin existence of dark matter
7 Gladys West mathematical modeling of the shape of the Earth which serv…
8 Flossie Wong-Staal first scientist to clone HIV and create a map of its genes…
9 Jennifer Doudna one of the primary developers of CRISPR, a ground-breaking…El objetivo es unir los data frames para obtener la información combinada de todas las columnas relevantes.
Joining with `by = join_by(name)`
Joining with `by = join_by(name)`# A tibble: 10 × 5
name profession birth_year death_year known_for
<chr> <chr> <dbl> <dbl> <chr>
1 Ada Lovelace Mathematician NA NA first co…
2 Marie Curie Physicist and Chemist NA NA theory o…
3 Janaki Ammal Botanist 1897 1984 hybrid s…
4 Chien-Shiung Wu Physicist 1912 1997 confim a…
5 Katherine Johnson Mathematician 1918 2020 calculat…
6 Rosalind Franklin Chemist 1920 1958 <NA>
7 Vera Rubin Astronomer 1928 2016 existenc…
8 Gladys West Mathematician 1930 NA mathemat…
9 Flossie Wong-Staal Virologist and Molecular … 1947 NA first sc…
10 Jennifer Doudna Biochemist 1964 NA one of t…names(professions)[1] "name" "profession"names(dates)[1] "name" "birth_year" "death_year"names(works)[1] "name" "known_for"nrow(professions)[1] 10nrow(dates)[1] 8nrow(works)[1] 96.5.1 Tipos de uniones (joins)
6.5.1.1 left_join()
left_join(professions, dates)Joining with `by = join_by(name)`# A tibble: 10 × 4
name profession birth_year death_year
<chr> <chr> <dbl> <dbl>
1 Ada Lovelace Mathematician NA NA
2 Marie Curie Physicist and Chemist NA NA
3 Janaki Ammal Botanist 1897 1984
4 Chien-Shiung Wu Physicist 1912 1997
5 Katherine Johnson Mathematician 1918 2020
6 Rosalind Franklin Chemist 1920 1958
7 Vera Rubin Astronomer 1928 2016
8 Gladys West Mathematician 1930 NA
9 Flossie Wong-Staal Virologist and Molecular Biologist 1947 NA
10 Jennifer Doudna Biochemist 1964 NA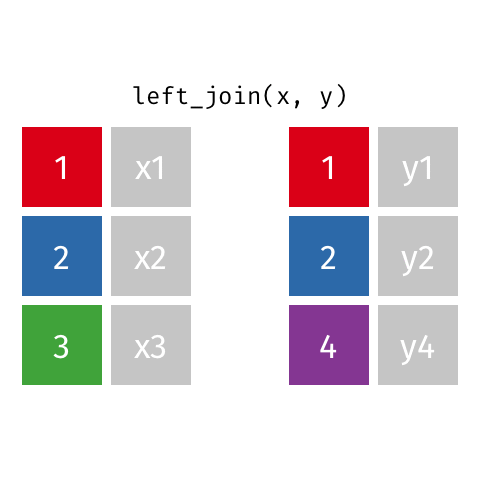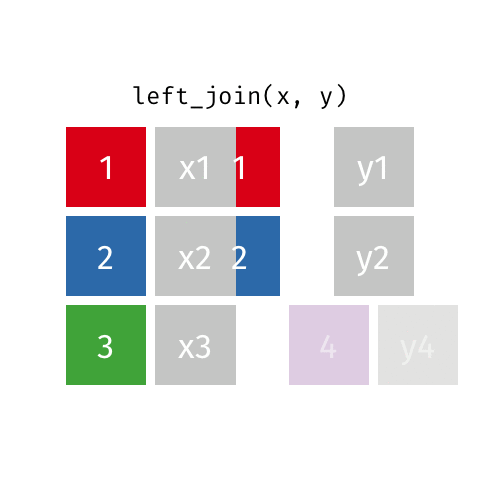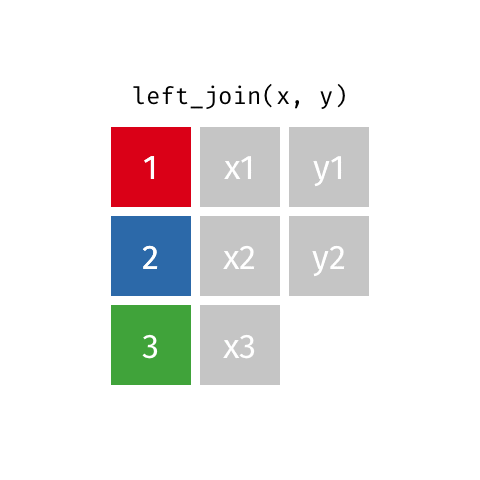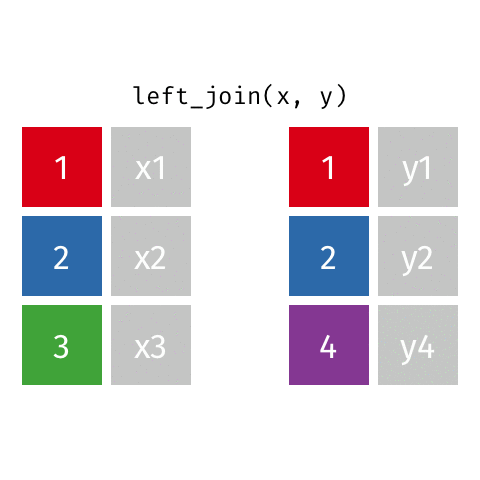
6.5.1.2 right_join()
right_join(professions, dates)Joining with `by = join_by(name)`# A tibble: 8 × 4
name profession birth_year death_year
<chr> <chr> <dbl> <dbl>
1 Janaki Ammal Botanist 1897 1984
2 Chien-Shiung Wu Physicist 1912 1997
3 Katherine Johnson Mathematician 1918 2020
4 Rosalind Franklin Chemist 1920 1958
5 Vera Rubin Astronomer 1928 2016
6 Gladys West Mathematician 1930 NA
7 Flossie Wong-Staal Virologist and Molecular Biologist 1947 NA
8 Jennifer Doudna Biochemist 1964 NA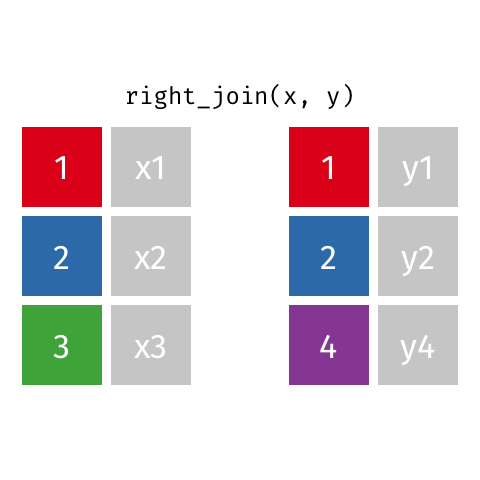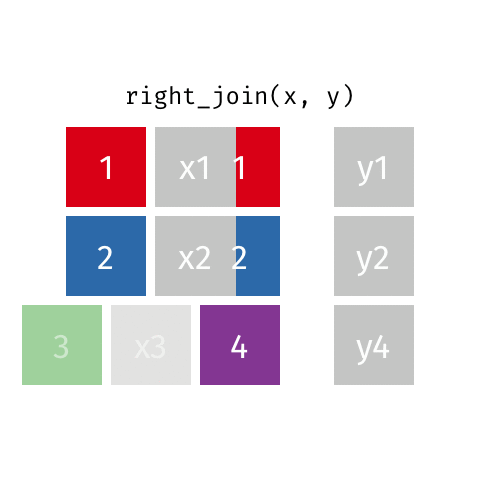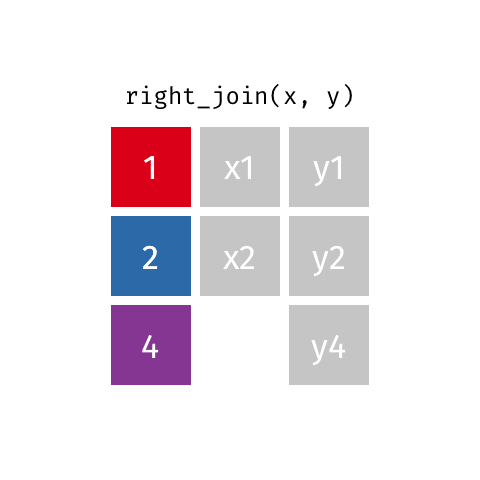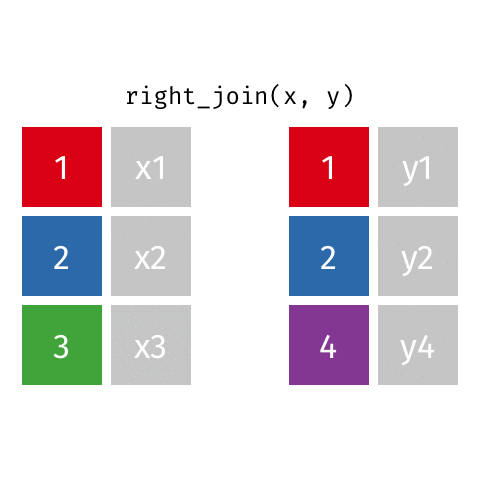
6.5.1.3 full_join()
full_join(professions, dates)Joining with `by = join_by(name)`# A tibble: 10 × 4
name profession birth_year death_year
<chr> <chr> <dbl> <dbl>
1 Ada Lovelace Mathematician NA NA
2 Marie Curie Physicist and Chemist NA NA
3 Janaki Ammal Botanist 1897 1984
4 Chien-Shiung Wu Physicist 1912 1997
5 Katherine Johnson Mathematician 1918 2020
6 Rosalind Franklin Chemist 1920 1958
7 Vera Rubin Astronomer 1928 2016
8 Gladys West Mathematician 1930 NA
9 Flossie Wong-Staal Virologist and Molecular Biologist 1947 NA
10 Jennifer Doudna Biochemist 1964 NA
6.5.1.4 inner_join()
inner_join(professions, dates)Joining with `by = join_by(name)`# A tibble: 8 × 4
name profession birth_year death_year
<chr> <chr> <dbl> <dbl>
1 Janaki Ammal Botanist 1897 1984
2 Chien-Shiung Wu Physicist 1912 1997
3 Katherine Johnson Mathematician 1918 2020
4 Rosalind Franklin Chemist 1920 1958
5 Vera Rubin Astronomer 1928 2016
6 Gladys West Mathematician 1930 NA
7 Flossie Wong-Staal Virologist and Molecular Biologist 1947 NA
8 Jennifer Doudna Biochemist 1964 NA
6.5.1.5 semi_join()
semi_join(professions, dates)Joining with `by = join_by(name)`# A tibble: 8 × 2
name profession
<chr> <chr>
1 Janaki Ammal Botanist
2 Chien-Shiung Wu Physicist
3 Katherine Johnson Mathematician
4 Rosalind Franklin Chemist
5 Vera Rubin Astronomer
6 Gladys West Mathematician
7 Flossie Wong-Staal Virologist and Molecular Biologist
8 Jennifer Doudna Biochemist 6.5.1.5.1 Ejemplo visual de semi_join()
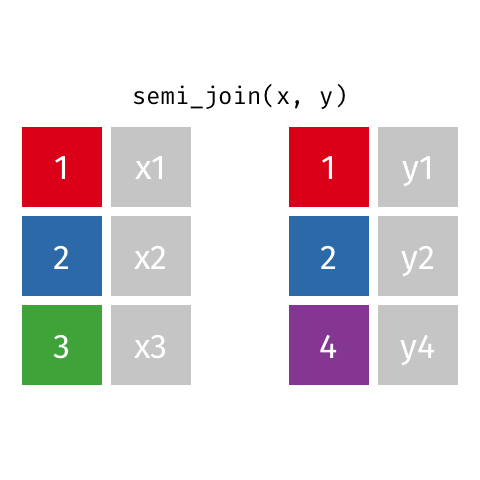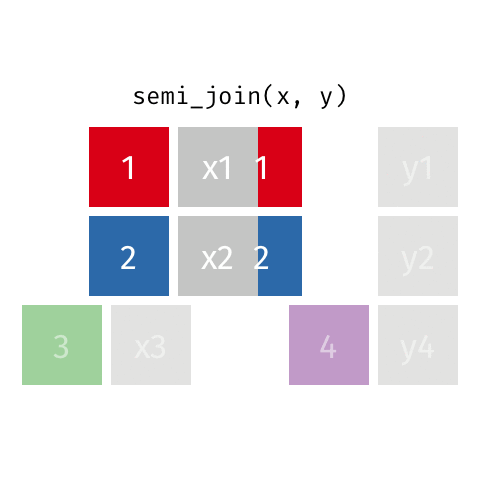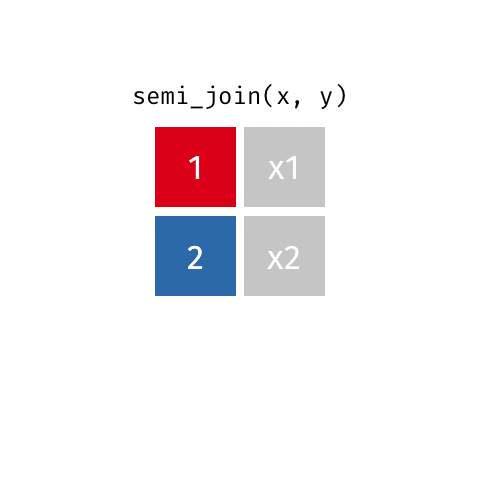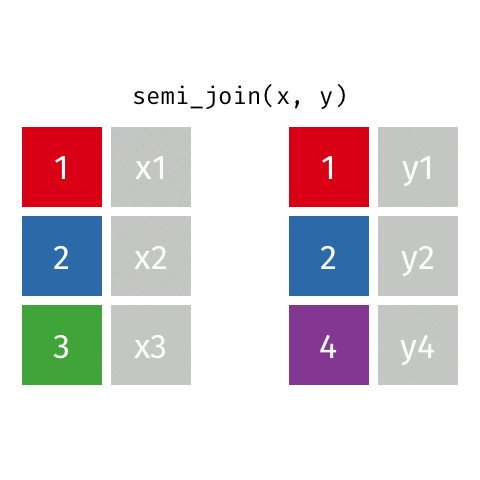
6.5.1.6 anti_join()
anti_join(professions, dates)Joining with `by = join_by(name)`# A tibble: 2 × 2
name profession
<chr> <chr>
1 Ada Lovelace Mathematician
2 Marie Curie Physicist and Chemist
6.5.2 Ejemplo completo
professions %>%
left_join(dates) %>%
left_join(works)Joining with `by = join_by(name)`
Joining with `by = join_by(name)`# A tibble: 10 × 5
name profession birth_year death_year known_for
<chr> <chr> <dbl> <dbl> <chr>
1 Ada Lovelace Mathematician NA NA first co…
2 Marie Curie Physicist and Chemist NA NA theory o…
3 Janaki Ammal Botanist 1897 1984 hybrid s…
4 Chien-Shiung Wu Physicist 1912 1997 confim a…
5 Katherine Johnson Mathematician 1918 2020 calculat…
6 Rosalind Franklin Chemist 1920 1958 <NA>
7 Vera Rubin Astronomer 1928 2016 existenc…
8 Gladys West Mathematician 1930 NA mathemat…
9 Flossie Wong-Staal Virologist and Molecular … 1947 NA first sc…
10 Jennifer Doudna Biochemist 1964 NA one of t…6.5.3 Estudio de caso: Registros de estudiantes
Tienes:
- Enrolment: Registros oficiales de matrícula de la universidad.
- Survey: Información proporcionada por los estudiantes, que incluye a los que llenaron la encuesta pero abandonaron la clase.
enrolment <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d08-multi-df/data/students/enrolment.csv")Rows: 3 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): name
dbl (1): id
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.survey <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d08-multi-df/data/students/survey.csv")Rows: 4 Columns: 3
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (2): name, username
dbl (1): id
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.enrolment# A tibble: 3 × 2
id name
<dbl> <chr>
1 1 Dave Friday
2 2 Hermine
3 3 Sura Selvarajahsurvey# A tibble: 4 × 3
id name username
<dbl> <chr> <chr>
1 2 Hermine bakealongwithhermine
2 3 Sura surasbakes
3 4 Peter peter_bakes
4 5 Mark thebakingbuddha Necesitas obtener la información completa de la clase
enrolment %>%
left_join(survey, by = "id")# A tibble: 3 × 4
id name.x name.y username
<dbl> <chr> <chr> <chr>
1 1 Dave Friday <NA> <NA>
2 2 Hermine Hermine bakealongwithhermine
3 3 Sura Selvarajah Sura surasbakes Además requires los estudiantes que no hicieron la encuesta
enrolment %>%
anti_join(survey, by = "id")# A tibble: 1 × 2
id name
<dbl> <chr>
1 1 Dave FridayEstudiantes que abandonaron la clase
survey %>%
anti_join(enrolment, by = "id")# A tibble: 2 × 3
id name username
<dbl> <chr> <chr>
1 4 Peter peter_bakes
2 5 Mark thebakingbuddha6.5.4 Estudio de caso: Ventas de supermercado
Tienes:
- Purchases: Una fila por cliente por ítem, listando las compras realizadas.
- Prices: Una fila por ítem en la tienda, con los precios correspondientes.
purchases <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d08-multi-df/data/sales/purchases.csv")Rows: 5 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): item
dbl (1): customer_id
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.prices <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d08-multi-df/data/sales/prices.csv")Rows: 5 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): item
dbl (1): price
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.purchases# A tibble: 5 × 2
customer_id item
<dbl> <chr>
1 1 bread
2 1 milk
3 1 banana
4 2 milk
5 2 toilet paperprices# A tibble: 5 × 2
item price
<chr> <dbl>
1 avocado 0.5
2 banana 0.15
3 bread 1
4 milk 0.8
5 toilet paper 3 Calcular el ingreso total
purchases %>%
left_join(prices) %>%
summarise(total_revenue = sum(price))Joining with `by = join_by(item)`# A tibble: 1 × 1
total_revenue
<dbl>
1 5.75Ingresos por cliente
purchases %>%
left_join(prices) %>%
group_by(customer_id) %>%
summarise(total_revenue = sum(price))Joining with `by = join_by(item)`# A tibble: 2 × 2
customer_id total_revenue
<dbl> <dbl>
1 1 1.95
2 2 3.8 6.6 Reestructuración de datos con tidyr
Ahora trabajaremos con la reestructuración de los datos, especialmente útil cuando trabajamos con datos de series temporales o de diferentes categorías. El concepto clave es Pivotar.
>Existen tres reglas interrelacionadas que hacen que un conjunto de datos sea ordenado:
- Cada variable debe tener su propia columna.
- Cada observación debe tener su propia fila.
- Cada valor debe tener su propia celda.
El objetivo de tidyr es ayudarte a organizar tus datos mediante:
- Transformación entre datos amplios y largos.
- División y combinación de columnas de caracteres.
- Anidamiento y desanidamiento de columnas.
- Clarificación de cómo tratar los valores
NA.

Usaremos estos datos para ver este concepto
customers <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d09-tidying/data/sales/customers.csv")
prices <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d09-tidying/data/sales/prices.csv")customers# A tibble: 2 × 4
customer_id item_1 item_2 item_3
<dbl> <chr> <chr> <chr>
1 1 bread milk banana
2 2 milk toilet paper <NA> Queremos transformar los datos de formato “ancho” a “largo” utilizando pivot_longer().
# A tibble: 6 × 3
customer_id item_no item
<dbl> <chr> <chr>
1 1 item_1 bread
2 1 item_2 milk
3 1 item_3 banana
4 2 item_1 milk
5 2 item_2 toilet paper
6 2 item_3 <NA> 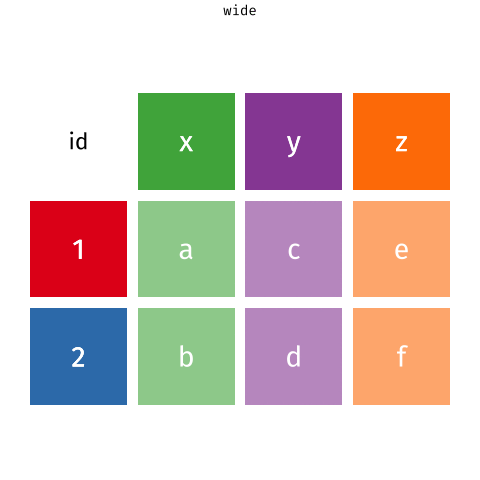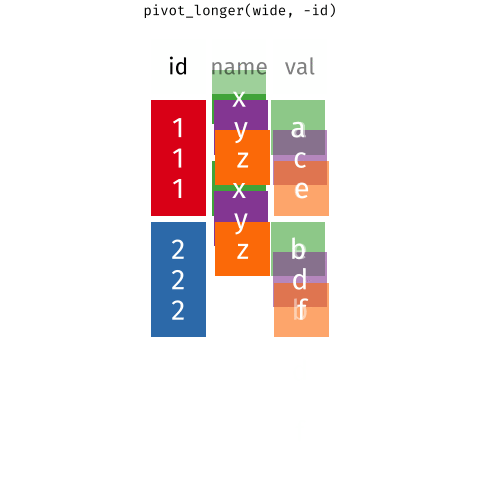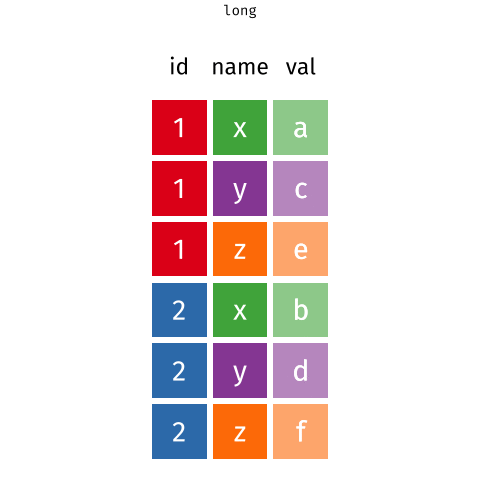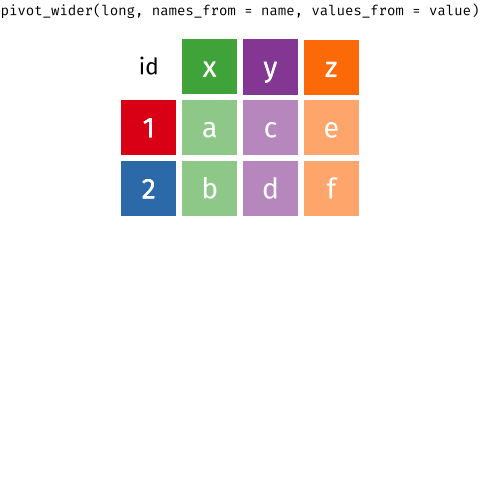
6.6.1 Datos anchos vs. largos
Datos más anchos (más columnas):
customers# A tibble: 2 × 4
customer_id item_1 item_2 item_3
<dbl> <chr> <chr> <chr>
1 1 bread milk banana
2 2 milk toilet paper <NA> Datos más largos (más filas):
customers %>%
pivot_longer(
cols = item_1:item_3,
names_to = "item_no",
values_to = "item"
)# A tibble: 6 × 3
customer_id item_no item
<dbl> <chr> <chr>
1 1 item_1 bread
2 1 item_2 milk
3 1 item_3 banana
4 2 item_1 milk
5 2 item_2 toilet paper
6 2 item_3 <NA> 6.6.2 Usando pivot_longer()
Sintaxis básica de pivot_longer()
pivot_longer(
data,
cols,
names_to = "name",
values_to = "value"
)Ejemplo práctico con pivot_longer()
purchases <- customers %>%
pivot_longer(
cols = item_1:item_3,
names_to = "item_no",
values_to = "item"
)
purchases# A tibble: 6 × 3
customer_id item_no item
<dbl> <chr> <chr>
1 1 item_1 bread
2 1 item_2 milk
3 1 item_3 banana
4 2 item_1 milk
5 2 item_2 toilet paper
6 2 item_3 <NA> 6.6.3 ¿Por qué pivotar?
Pivotamos los datos principalmente porque el siguiente paso de nuestro análisis lo necesita.
prices# A tibble: 5 × 2
item price
<chr> <dbl>
1 avocado 0.5
2 banana 0.15
3 bread 1
4 milk 0.8
5 toilet paper 3 purchases %>%
left_join(prices)Joining with `by = join_by(item)`# A tibble: 6 × 4
customer_id item_no item price
<dbl> <chr> <chr> <dbl>
1 1 item_1 bread 1
2 1 item_2 milk 0.8
3 1 item_3 banana 0.15
4 2 item_1 milk 0.8
5 2 item_2 toilet paper 3
6 2 item_3 <NA> NA Pivotar de nuevo a datos amplios
purchases %>%
pivot_wider(
names_from = item_no,
values_from = item
)# A tibble: 2 × 4
customer_id item_1 item_2 item_3
<dbl> <chr> <chr> <chr>
1 1 bread milk banana
2 2 milk toilet paper <NA> 6.6.4 Estudio de caso: Aprobación de Donald Trump

Fuente: FiveThirtyEight
Datos
trump <- read_csv("https://raw.githubusercontent.com/tidyverse/datascience-box/main/course-materials/_slides/u2-d09-tidying/data/trump/trump.csv")Rows: 2702 Columns: 4
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): subgroup
dbl (2): approval, disapproval
date (1): date
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.trump# A tibble: 2,702 × 4
subgroup date approval disapproval
<chr> <date> <dbl> <dbl>
1 Voters 2020-10-04 44.7 52.2
2 Adults 2020-10-04 43.2 52.6
3 Adults 2020-10-03 43.2 52.6
4 Voters 2020-10-03 45.0 51.7
5 Adults 2020-10-02 43.3 52.4
6 Voters 2020-10-02 44.5 52.1
7 Voters 2020-10-01 44.1 52.8
8 Adults 2020-10-01 42.7 53.3
9 Adults 2020-09-30 42.2 53.7
10 Voters 2020-09-30 44.2 52.7
# ℹ 2,692 more rowsTransformar los datos para crear un gráfico que muestre la aprobación y desaprobación a lo largo del tiempo, diferenciando entre adultos y votantes registrados.
trump_longer <- trump %>%
pivot_longer(
cols = c(approval, disapproval),
names_to = "rating_type",
values_to = "rating_value"
)
trump_longer# A tibble: 5,404 × 4
subgroup date rating_type rating_value
<chr> <date> <chr> <dbl>
1 Voters 2020-10-04 approval 44.7
2 Voters 2020-10-04 disapproval 52.2
3 Adults 2020-10-04 approval 43.2
4 Adults 2020-10-04 disapproval 52.6
5 Adults 2020-10-03 approval 43.2
6 Adults 2020-10-03 disapproval 52.6
7 Voters 2020-10-03 approval 45.0
8 Voters 2020-10-03 disapproval 51.7
9 Adults 2020-10-02 approval 43.3
10 Adults 2020-10-02 disapproval 52.4
# ℹ 5,394 more rowsggplot(
trump_longer,
aes(x = date, y = rating_value, color = rating_type, group = rating_type)
) +
geom_line() +
facet_wrap(~subgroup)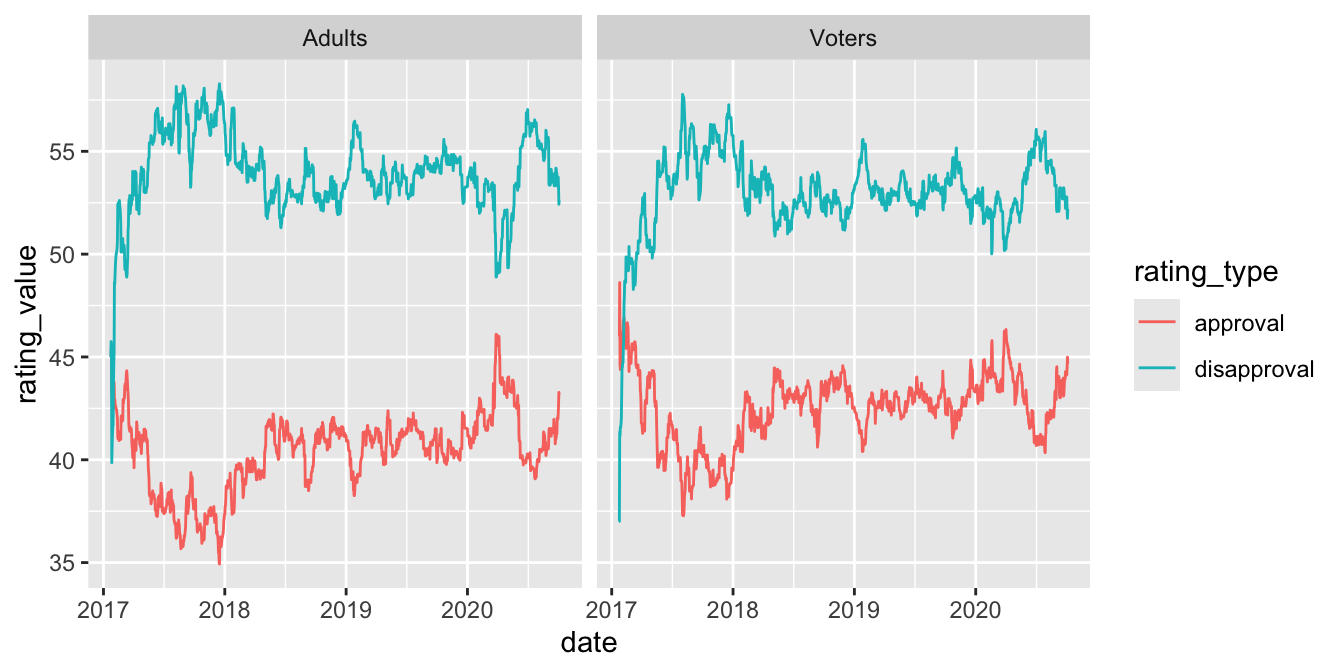
Versión mejorada
ggplot(trump_longer, aes(x = date, y = rating_value, shape = rating_type)) +
geom_point(size=1) +
cowplot::theme_cowplot()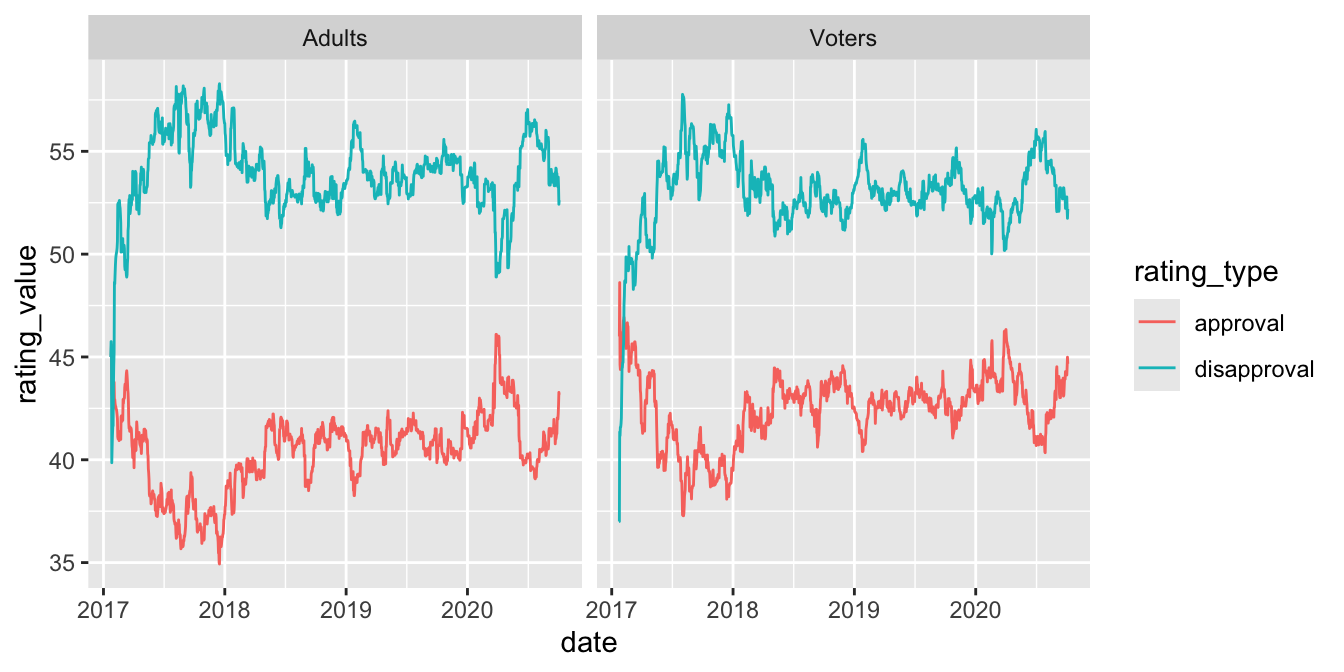
trump_labels <- trump_longer |>
group_by(rating_type) |>
arrange(desc(date)) |>
slice(1) |>
ungroup()
trump_labels_approval <- trump_labels |>
filter(rating_type == "approval")
trump_labels_disapproval <- trump_labels |>
filter(rating_type == "disapproval")
ggplot(trump_longer, aes(x = date, y = rating_value, color = rating_type)) +
geom_line() +
# scale_color_brewer(palette = "Set2")+
scale_color_manual(values = c( "disapproval" = "black", "approval" = "red" ))+
labs(
x = "Fecha",
y = "Aprobación",
title = "Popularidad de Trump",
subtitle = "Puntajes recolectados en encuestas",
caption = "Elaboración propia.",
color = ""
) +
annotate("label",
x = trump_labels_disapproval$date ,
y = trump_labels_disapproval$rating_value,
label = "Desaprobación")+
annotate("label", x= as.Date("2019-01-01"), y = 60 , label = "Aprobación")+
cowplot::theme_cowplot()+
theme(legend.position = "none")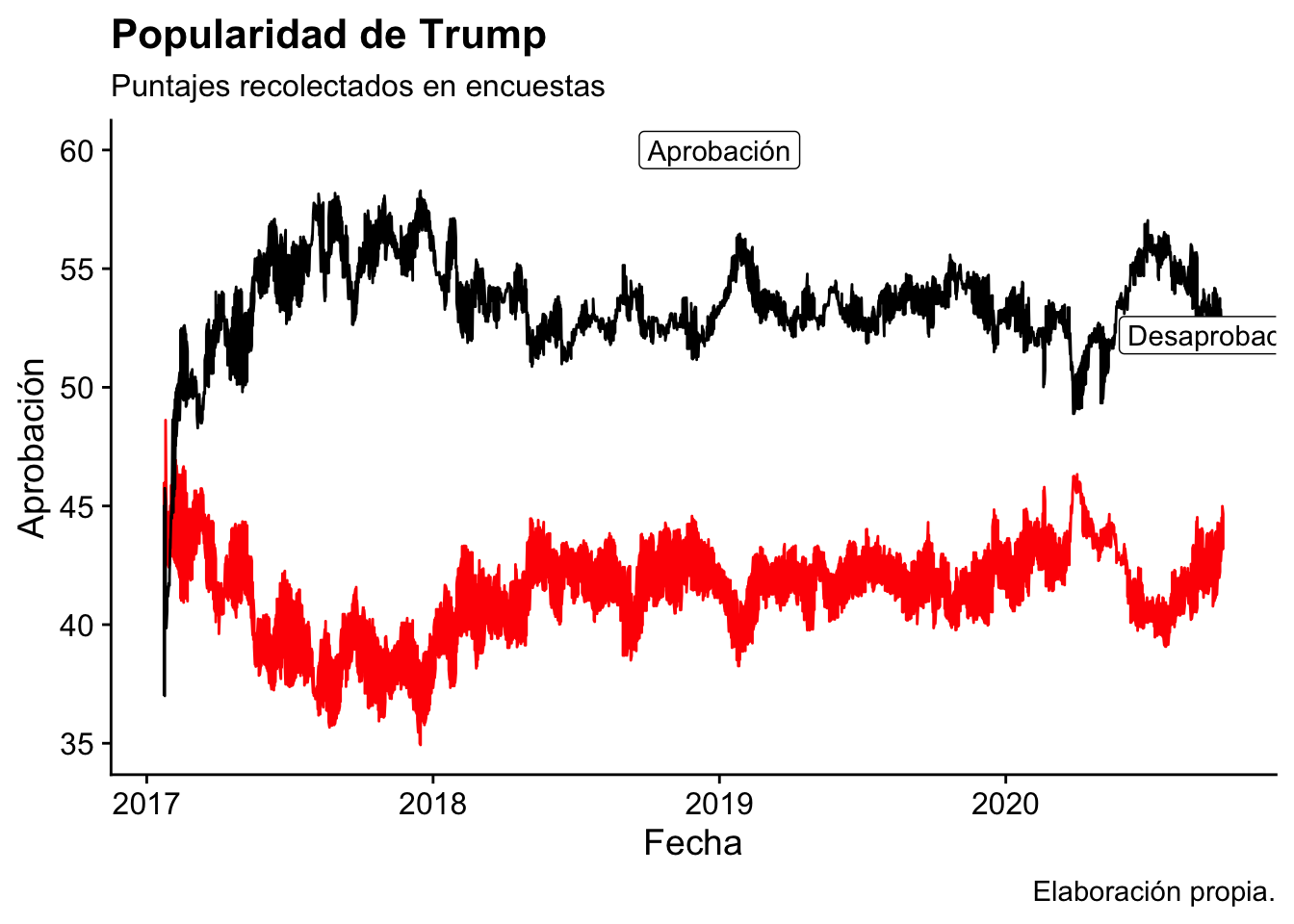
6.7 Carrera ggplot
En todos los ejemplos, les sugiero seguir los siguientes pasos:
Exploración de Datos: Carguen el dataset y explora brevemente su estructura para identificar las variables clave.
Análisis: Analiza la pregunta planteada. Entienda bien el contexto y la relación entre las variables.
- ¿Qué estás tratando de averiguar?
- ¿Qué variables son importantes para tu análisis?
- ¿Qué tipo de gráfico sería más adecuado para visualizar los datos?
Limpiar y Preparar los Datos: Limpia y prepara los datos según sea necesario para el análisis. Te recomiendo que revises estas funciones en
tidyverse:mutate()parse_number()separate()pivot_longer()
Visualización: Con
ggplot2crea el gráfico que mejor represente los datos y responda a la pregunta planteada.ggplot()geom_point()geom_line()geom_bar()facet_wrap()labs()theme()
Estilo: Mejora la visualización con colores, etiquetas y temas adecuados. Recuerde que menos es más.
6.7.1 Desafío 1: Análisis de adopción de funcionalidades
Imagina que eres un analista en una empresa de marketing digital. En 2015 se introdujo una nueva funcionalidad, denominada “Funcionalidad Z”, que permite a los clientes de tu compañía crear mejores anuncios e introduce una nueva fuente de ingresos para tu plataforma. El desafío es que “Funcionalidad Z” tiene una curva de aprendizaje elevada, por lo que ha sido complicado para los clientes utilizarla. Sin embargo, se ha observado una mejora en el tiempo en términos de uso de la “Funcionalidad Z” y el aumento de ingresos derivados de esta. En una reciente reunión sobre este tema, el jefe de soporte al cliente planteó una pregunta sobre cómo se ve la adopción de “Funcionalidad Z” específicamente en los nuevos anunciantes, aquellos que crean un anuncio en tu plataforma por primera vez.
Datos
6.7.2 Desafío 2: Análisis de las tasas de diabetes y crecimiento en centros médicos
Eres un analista que trabaja para un gran sistema de atención médica con varios centros médicos en diferentes estados. Tu rol es analizar las tendencias en la base de pacientes y comunicar tus hallazgos para ayudar a los administradores a tomar decisiones organizacionales. Recientemente, has identificado un aumento en las tasas de diabetes en todos los centros médicos (A-M) en una región. Usando un modelo en particular hiciste una proyección desde el 2020 al 2023 de la tendencia de crecimiento. Si esta tendencia continúa, los centros podrían no contar con suficiente personal para atender a los pacientes de manera adecuada. En particular, descubriste que para el 2023 habrían 14 000 nuevos pacientes por año en los próximos 4 años. Estás trabajando en una presentación que compartirá estos hallazgos con los administradores del centro médico.
Debes considerar lo siguiente:
- Antes de realizar cualquier análisis, piensa en tu audiencia, que es un administrador senior en el sistema de atención médica. ¿Cuáles son sus preocupaciones principales? Haz una lista de factores que podrían preocupar a alguien en su posición.
- Ejemplo: La preocupación podría incluir la falta de personal, aumento de costos, o un sistema incapaz de manejar el aumento en la cantidad de pacientes.
- Define cuál será el mensaje principal que deseas comunicar. ¿Qué deberían recordar los administradores después de tu presentación? Haz suposiciones si es necesario, pero elabora un mensaje claro y directo.
- Ejemplo de “Gran Idea”: “La rápida tasa de aumento en los pacientes con diabetes requiere una asignación urgente de recursos adicionales en todos los centros médicos para prevenir la sobrecarga del sistema.”
- Piensa en la estructura de la historia que vas a contar. Identifica el “conflicto” que enfrenta tu audiencia (ejemplo: aumento de pacientes) y cómo tus datos proporcionan una solución (ejemplo: asignación de más recursos). Organiza tu contenido en una narrativa clara con puntos clave.
- Ejemplo:
- Conflicto: Aumento de la tasa de pacientes con diabetes.
- Solución: Predicción y planificación para recursos adicionales.
- Piezas clave de contenido: Visualización de los datos actuales, proyección de las tasas futuras, plan de acción para asignación de recursos.
- Ejemplo:
Datos